(2: 农业部淡水渔业健康养殖重点实验室,浙江省淡水水产研究所,湖州 313001)
(2: Key Laboratory of Fish Health and Nutrition of Zhejiang Province, Zhejiang Institute of Freshwater Fisheries, Huzhou 313001, P.R.China)
河川沙塘鳢(Odontobutis potamophila)隶属于鲈形目(Perciformes)、鰕虎鱼亚目(Gobioidei)、塘鳢科(Eleotridae)、沙塘鳢属(Odontobutis),是一种较为名贵的小型经济鱼类,主要分布于长江中、下游及沿江各支流,钱塘江水系,闽江水系,偶见于黄河水系[1].该鱼个体虽小,但含肉量高、肉质鲜嫩、营养价值高,在江、浙、沪等地区历来被视为名贵鱼类,深受消费者喜爱[2].近年来,价格更是一路攀升,已成为一种极具发展潜力的水产养殖新品种.
近年来随着DNA分子技术的快速发展,从分子水平研究鱼类物种间的亲缘关系及进行物种鉴定已成为可能.线粒体DNA(Mitochondrial DNA,mtDNA)结构简单,由于呈母性遗传特点,复制过程中缺乏修复机制,导致序列的累积变异速率远远高于核基因组的变异速率,进化速度快且不发生重组,已成为群体遗传学和系统发育等研究的理想分子标记[3-4].细胞色素b(cyt b)基因为mtDNA上的蛋白质编码基因,进化速度适中,且容易使用一些通用引物进行扩增和测序,因此适合进行种间到种内水平上的系统发生研究,在鱼类群体遗传结构与系统发育关系中有着广泛的应用[5-7].目前,通过线粒体DNA不同区域和基因序列对沙塘鳢种群的遗传结构和系统演化关系已经进行了一些研究.谢楠等[8-9]对河川沙塘鳢线粒体cyt b基因序列分析,并与塘鳢科其余种类进行比对,构建鳢科鱼类系统发育树.郁建锋等[10]对太湖流域河川沙塘鳢线粒体12S和16S rRNA基因序列进行分析,表明太湖流域河川沙塘鳢有别于其他类别的沙塘鳢,且与已知的河川沙塘鳢存在分子遗传进化差异.李妍等[11]采用简单序列重复区间扩增多态性(ISSR)分子标记技术对河川沙塘鳢2个群体进行遗传分析,表明2个群体的遗传分化已经达到种群分化水平.侯新远等[12]基于线粒体D-loop基因序列研究我国5种虾虎鱼类的亲缘关系,而目前对长江水系内部不同湖泊和流域的河川沙塘鳢遗传结构分析还未见报道.本研究采集长江中下游鄱阳湖、巢湖、大纵湖、固城湖、太湖5个湖泊的河川沙塘鳢种群,通过mtDNA cyt b基因核苷酸序列的比较分析,探讨5个湖泊的河川沙塘鳢种群遗传多样性和进化关系,以深入了解5个湖泊河川沙塘鳢种群的遗传背景和种质资源现状,以利于在沙塘鳢育种中更好地发掘其遗传潜力,为沙塘鳢多性状复合育种技术的研究及新品系选育积累基础数据.
1 材料和方法 1.1 研究材料实验所用野生河川沙塘鳢(Odontobutis potamophila)采集于5个湖泊,分别是长江流域的鄱阳湖、巢湖和太湖,江苏省南京高淳的固城湖以及盐城市的大纵湖,每个种群采集样本数见表 1,剪取活鱼尾鳍,固定于无水乙醇中,放入4℃冰箱保存.
| 表 1 5个河川沙塘鳢种群的单倍型及遗传多样性参数 Tab.1 Parameter summary of haplotypes and genetic diversity of five populations of O. potamophila |
基因DNA提取参照Sambrook等[13]的方法.DNA浓度、质量分别用分光光度仪(Unico UV-4802H)和1 %琼脂糖凝胶电泳检测.总DNA稀释至100ng/μl,于-20℃储存备用.
1.3 PCR扩增及测序cyt b扩增和测序的引物为通用引物L14724和H15915[14],引物序列为L14724:5′-GACTTGAAAAACCACCGTTG-3′;H15915:5′-CTCCGATCTCCGGATTACAAGAC-3′,引物由生工生物工程(上海)股份有限公司合成.PCR反应体系为50μl:模板DNA 50ng;2×PCR Mix 25μl(包含Taq酶2.5U,dNTPs 10μmol,MgCl20.1 mmol),上、下游引物(10μmol/L)各1μl,其余体积用双蒸水补足.反应程序为:94℃预变性2min,94℃变性45s,60℃退火1min,72℃延伸1min,经35个循环后再72℃延伸10min.PCR产物经割胶纯化后,直接送至南京思普金生物科技有限公司利用正反引物进行双向测序.
1.4 数据分析DNA序列用BioEdit 7.0.5[15]软件比对剪切序列,MEGA 4.1软件[16]计算碱基含量,并构建系统进化树.通过DnaSP软件[17]计算变异位点,单倍型数、单倍型多样性指数(h)、平均核苷酸差异数(K)和核苷酸多样性指数(π)等,用Arlequin 3.01软件[18]中的Kimura 2-Parameters方法构建5个湖泊河川沙塘鳢种群间的相对遗传距离,用分子方差分析(AMOVA)方法计算遗传分化系数(Fst)并分析遗传变异来源与组成.
2 结果 2.1 序列特征经Bioedit软件对5个湖泊河川沙塘鳢种群115个个体的mtDNA cyt b序列进行比对剪切,获得1141bp的序列全长,并全部提交至GenBank,登录号为:KM924174~KM924288,MEGA 4.1软件分析115个序列的碱基组成平均为T(28.1 %)、C(28.8 %)、A(27.7 %)、G(15.4 %),A+T含量为55.8 %,略高于G+C含量(44.2 %).DnaSP软件分析显示,共有806个多态性位点,约占核苷酸总数的70.64 %,单态突变位点(singleton sites)163个,简约信息位点(parsimony informative sites)643个;同时还检测到344个插入或缺失位点,碱基插入或缺失主要以C或G单核苷酸重复片段的形式进行.115个个体中共检出87种单倍型序列.5个种群的平均h值为0.969±0.012,π为0.20081±0.00742(表 1).5个湖泊河川沙塘鳢种群中,大纵湖和鄱阳湖种群的h最高,固城湖种群的π最高,而太湖种群的h和π均最低.
2.2 群体变异和遗传结构将测序获得的115个个体的mtDNA序列输入Arlequin软件,使用Kimura 2-Parameters方法构建5个湖泊种群间的相对遗传距离和Fst(表 2).从遗传距离来看,巢湖种群与大纵湖种群间的遗传距离最远(0.424),遗传距离最近的是大纵湖种群与太湖种群(0.137);种群内部遗传距离从大到小依次为:固城湖(0.237)>大纵湖(0.189)>鄱阳湖(0.143)>巢湖(0.092)>太湖(0.056).5个种群的平均Fst为0.531,5个湖泊河川沙塘鳢种群间存在显著性遗传变异(P<0.05).
| 表 2 5个河川沙塘鳢种群间Kimura 2-Parameters遗传距离和Fst Tab.2 The average Kimura-2-Parameters distance and Fst among five O. potamophila populations |
为进一步分析种群间的相互遗传差别,通过Arlequin软件中AMOVA法估算5个种群间的遗传变异结构及来源.AMOVA结果显示,5个河川沙塘鳢种群内和种群间均具有很高的遗传变异.种群间的变异占总变异的53.15 %,而种群内的变异占总变异的46.85 %(表 3).分析单倍型数据所得遗传分化系数(Gst)为0.07743,基因流(Nm)为2.98;而115个个体的Gst为0.47076,Nm为0.28.
| 表 3 5个河川沙塘鳢种群间遗传差异的分子方差分析 Tab.3 Analysis of molecular variance among five populations of O. potamophila |
将115个河川沙塘鳢个体的mtDNA cyt b序列输入MEGA 4.1软件,对5个湖泊种群进行聚类分析,得到UPGMA聚类树(图 1).5个河川沙塘鳢地理种群没有明显聚类,形成了单系类群,太湖与大纵湖、固城湖种群首先聚为一支,接着与巢湖、鄱阳湖种群聚成的一支汇合.各单倍型存在相互散布在不同地理种群中的现象.
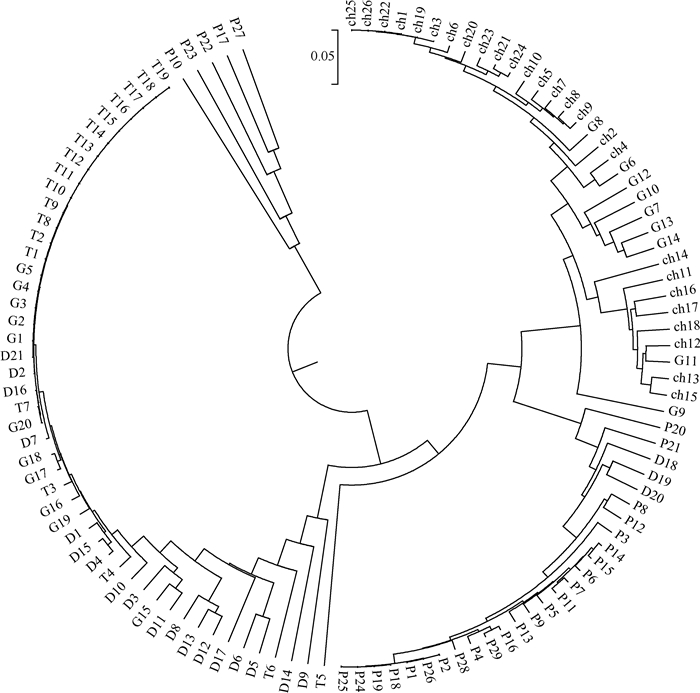
|
图 1 5个河川沙塘鳢种群的UPGMA聚类树 Fig.1 Dendrogram of five populations of O. potamophila by UPGMA method |
用Fu[19]的Fs中性检验检验5个湖泊河川沙塘鳢种群的显著偏离中性突变:Fs>0,表明种群趋于稳定;Fs<0,表明种群趋于扩张.结果显示,种群的整体Fs=1.122(P>0.05),表明5个河川沙塘鳢种群整体上趋于稳定,同时巢湖、固城湖、鄱阳湖和太湖种群的Fs>0,而大纵湖种群Fs<0.
3 讨论 3.1 遗传多样性遗传多样性的研究是生物多样性研究的重要内容,只有通过遗传多样性的研究才能从本质上揭示物种多样性的起源、变异和进化.本研究中5个湖泊河川沙塘鳢种群cyt b序列的碱基组成为T(28.1 %)、C(28.8 %)、A(27.7 %)、G(15.4 %),其中G含量显著低于其他碱基含量,表现出明显的反G偏倚,显示出cyt b基因的共同特征[20],同时这也是线粒体DNA的一个特点[21].本研究中河川沙塘鳢A+T含量为55.8 %,略高于G+C含量(44.2 %),与谢楠等[9]关于河川沙塘鳢cyt b基因序列的分析结果一致.
在5个湖泊河川沙塘鳢种群中的115个个体共检测出87个单倍型,表明河川沙塘鳢群体存在丰富的mtDNA多态性.与其他淡水鱼类(黄颡鱼[22]、乌鳢[23]、淮河野生鲇鱼[24])相比,河川沙塘鳢平均h和π相对较高(h=0.969±0.012,π=0.20081±0.00742),显示较高的遗传多样性,同时符合Grant等[25]提出的鱼类不同单倍型多样性与核苷酸多样性间组合的第2种类型,即高h和低π.而河川沙塘鳢表现出较高h、低π特征,这可能是由于河川沙塘鳢生命周期短,使其能在较短时间内积累较多的变异,表现出较高的单倍型多样性,但短时间内又不能积累足够的核苷酸多样性,从而导致种群核苷酸多样性较低[26].
5个湖泊中太湖种群河川沙塘鳢的h和π均为最低,这可能是由于太湖流域工业发达、污染严重,蓝藻水华暴发导致太湖水质恶化,鱼类大量死亡,河川沙塘鳢喜栖息于水质良好的水域中,环境污染破坏其栖息地,也势必导致群体急剧下降.同时为了治理太湖水质,太湖的各个入湖和出湖水道上都修建了大量的水利设施,河川沙塘鳢种群也处于隔离状态,造成太湖流域河川沙塘鳢群体遗传多样性丧失.
3.2 种群遗传结构与基因流本研究中,AMOVA对遗传变异的分析表明,全部遗传变异的53.15 %来自种群间,46.85 %来自种群内部,说明河川沙塘鳢各种群间以及各种群内个体间的遗传多样性均较高.种群间的遗传距离以及Fst是衡量种群多态程度的重要指标.二者的值越大,种群多态性程度越高[27].本研究中,河川沙塘鳢的遗传距离在0.137~0.424之间,根据遗传距离对物种的分类依据表明河川沙塘鳢无论是群体内还是群体间序列差异都较大[28-29],整体上反映了各地理种群之间亲缘关系较远,这也与AMOVA对遗传变异的分析结果一致.而5个湖泊河川沙塘鳢种群间的Fst为0.531,说明5个河川沙塘鳢种群间存在较高的遗传分化[30].种群间的遗传距离分析与Fst的分析结果相一致.
本研究中单倍型之间的Nm为2.98,单倍型Gst为0.07743,115个个体的Nm为0.28,Gst为0.47076,表明5个湖泊种群间存在基因流动较小,这可能与河川沙塘鳢的生活习性相关,河川沙塘鳢的游泳能力较弱,可能会导致群体间的基因交流较少.这与吕国庆的研究一致,认为种群之间的遗传变异程度也与其生态习性及生活史密切相关[31].
本研究结果也与李妍等[11]采用ISSR分子标记技术对江苏常熟河川沙塘鳢个体大小不等的2个群体进行遗传分析的结果类似,江苏常熟河川沙塘鳢2个群体的遗传分化已经达到种群分化水平.同时郁建锋等[10]通过分析太湖流域河川沙塘鳢线粒体12S和16S rRNA基因序列分析表明,在12S、16S rRNA基因水平上太湖流域河川沙塘鳢与其他沙塘鳢甚至是其他地区的河川沙塘鳢之间存在分子差异,这可能是与沙塘鳢长期的区域化生长繁殖有关,造成太湖流域河川沙塘鳢12S、16S rRNA基因不同程度的变异、分化,并被积累下来.
5个湖泊河川沙塘鳢种群UPGMA聚类树分析表明,5个地理种群没有明显聚类,形成了单系类群,各单倍型存在相互散布在不同地理种群中的现象,这可能是由于本实验受样本数的局限,种群遗传多样性和种群进化的具体参数等有待于今后进一步的研究.此外,仅靠mtDNA cyt b基因来评估河川沙塘鳢不同种群的遗传多样性和特征是远远不够的,要深入了解其遗传背景还需要综合运用多种分子标记手段(如简单序列重复(SSR)、扩增酶切片断多态性(AFLP)、单核苷酸多态性(SNP)等)进一步研究河川沙塘鳢的遗传多样性及其适应机制.
河川沙塘鳢是我国一种传统的名贵食用鱼类,在我国广泛分布于长江中下游、钱塘江、闽江等水系,具有一定的渔业价值.近年来,由于自然资源过度利用、天然水域环境污染加剧等因素,沙塘鳢自然产量明显减少,据本课题组2008—2010年调查,江苏省苏北地区水域中已很难捕获,并且有趋于灭绝的危险.本研究对河川沙塘鳢mtDNA cyt b基因的遗传背景、遗传结构及变异水平进行探讨,为河川沙塘鳢的种质资源评估提供基础材料.本研究表明,5个湖泊的河川沙塘鳢种群具有较高的遗传变异,过度捕捞、生态环境的破坏以及人工繁殖数代退化后的种苗流入自然水域,加之河川沙塘鳢的基因流动较少,会造成野生资源的进一步退化.因此,急需加强对河川沙塘鳢野生种质资源的保护,建立规范的良种保种和选育基地,在保证种群自然扩张、提高遗传多样性的同时,保护种质资源,维护良种的纯度和遗传稳定性,还能大量供应市场,满足广大人民群众的消费需求.
| [1] |
伍汉霖, 吴小清, 解玉浩. 中国沙塘鳢属鱼类的整理和一新种的叙述. 上海水产大学学报, 1993, 2(1): 52-61. |
| [2] |
胡先成, 孙帼英. 河川沙塘鳢消化系统的发育及仔、稚、幼鱼摄食的研究. 上海水产大学学报, 1996, 5(2): 75-82. |
| [3] |
Brown WM. Evolution of animal mitochondrial DNA. In: Nei M, Koehn RK eds. Evolution of genes and proteins. Sinauer: Sun-derland MA, 1983: 62-88.
|
| [4] |
Saccone C, Pesole G, Sbisa E. The main regulatory region of mammalian mitochondrial DNA: structure-function model and evolutionary pattern. Journal of Molecular Evolution, 1991, 33(1): 83-91. DOI:10.1007/BF02100199 |
| [5] |
彭作刚, 张耀光, 何舜平等. 从细胞色素b基因序列变异分析中国鲇形目鱼类的系统发育. 遗传学报, 2005, 32(2): 145-154. |
| [6] |
陈合格, 刘文彬, 李建中等. 三种鳖线粒体DNA细胞色素b基因序列的比较分析. 水生生物学报, 2006, 30(4): 380-385. |
| [7] |
程起群, 温俊娥, 王云龙等. 刀鲚与湖鲚线粒体细胞色素b基因片段多态性及遗传关系. 湖泊科学, 2006, 18(4): 425-430. DOI:10.18307/2006.0416 |
| [8] |
谢楠, 冯晓宇, 郭水荣等. 河川沙塘鳢(Odontobutis potamophila)线粒体细胞色素b(Cyt b)基因序列分析. 杭州农业与科学, 2010, 2: 22-26. |
| [9] |
谢楠, 冯晓宇, 郭水荣等. 河川沙塘鳢线粒体细胞色素b(Cyt b)基因序列分析. 动物科学, 2010, 19: 288-290. |
| [10] |
郁建锋, 韩晓磊, 郭倩林等. 太湖流域河川沙塘鳢线粒体12S和16S rRNA基因序列分析. 江苏农业科学, 2012, 40(12): 48-51. DOI:10.3969/j.issn.1002-1302.2012.12.016 |
| [11] |
李妍, 荣楠, 徐建荣等. 河川沙塘鳢的ISSR分析. 安徽农业科学, 2010, 38(2): 606-608. |
| [12] |
侯新远, 祝斐, 张丽娟等. 基于线粒体D-loop基因序列研究我国5种虾虎鱼类的系统进化关系. 海洋渔业, 2013, 35(1): 1-7. |
| [13] |
Sambrook J, Russell DW. Molercular cloning: A laboratory mamual(3rd edition). New York: Cold Spring Harkbor Labroatory Press, 2002, 463-469.
|
| [14] |
Xiao WH, Zhang YP, Liu HZ. Molecular systematics of Xenocyprinae(Teleostei: Cyprinidae): Taxonomy, biogeography and coevolution of a special group restricted in East Asia. Molecular Phylogenetics and Evolution, 2001, 18(2): 163-173. DOI:10.1006/mpev.2000.0879 |
| [15] |
Hall T. BioEdit v. 7. 0. 5. Biological sequence alignment editor for windows. Ibis Therapeutics a division of Isis pharmaceuticals. http://www.mbio.nesu.edu/bioefit.html, 2005.
|
| [16] |
Tamura K, Dudley J, Nei M et al. MEGA4: Molecular evolutionary genetics analysis(MEGA)software version 4. 0. Molecular Biology and Evolution, 2007, 24(8): 1596-1599. DOI:10.1093/molbev/msm092 |
| [17] |
Rozas J, Anchez-DelBarrio JC, Esseguer XM et al. DnaSP, DNA polymorphism analyses by the coalescent and other methods. Bioinformatics, 2003, 19(18): 2496-2497. DOI:10.1093/bioinformatics/btg359 |
| [18] |
Excoffier L, Laval G, Schneider S. Arlequin ver 3. 01: An integrated software package for population genetics data analysis. Berne: Computational and Molecular Population Genetics Laboratory(CMPG). Switzerland: University of Berne, 2006.
|
| [19] |
Fu YX. Statistical tests of neutrality of mutations against population growth, hitchhiking and backgrounds selection. Genetics, 1997, 147(2): 915-925. |
| [20] |
Rogers AR, Harpending H. Population growth makes waves in the distribution of pairwise genetic differences. Molecular Biology and Evolution, 1992, 9(3): 552-569. |
| [21] |
Wolstenholme DR. Animal mitochondrial DNA: Structure and evolution. In: Wolstenholme DR ed. Mitochondrial genomes. San Diego: Academic Press, 1992: 173-372.
|
| [22] |
钟立强, 刘朋朋, 潘建林等. 长江中下游5个湖泊黄颡鱼(Pelteobagrus fulvidraco)种群线粒体细胞色素b基因的遗传变异分析. 湖泊科学, 2013, 25(2): 302-308. DOI:10.18307/2013.0219 |
| [23] |
肖明松, 崔峰, 康健等. 淮河乌鳢线粒体DNA控制区结构分析及遗传多样性研究. 华中师范大学学报:自然科学版, 2013, 47(1): 82-90. |
| [24] |
肖明松, 崔峰, 康健等. 淮河野生鲇鱼线粒体Cytb基因的序列变异与遗传结构分析. 动物学杂志, 2013, 48(1): 75-86. |
| [25] |
Grant WS, Bowen BW. Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation. Heredity, 1998, 89(5): 415-426. DOI:10.1093/jhered/89.5.415 |
| [26] |
Avise JC. Phylogeography: The history and formation of species. Cambridge: Harvard University Press, 2000, 20-147.
|
| [27] |
陈大庆, 张春霖, 鲁成等. 青海湖裸鲤繁殖群体线粒体基因组D-loop区序列多态性. 中国水产科学, 2006, 13(5): 800-806. |
| [28] |
根井正利. 分子群体遗传学与进化论. 北京: 农业出版社, 1975, 121-133.
|
| [29] |
Shaklee JB. Speciation and evolution of marine fishes studied by electrophoresis analysis of proteins. Pacific Science, 1982, 36(2): 141-157. |
| [30] |
吕宝忠, 钟扬, 高莉萍等. 分子进化与系统发育. 北京: 高等教育出版社, 2002.
|
| [31] |
吕国庆. 鱼类线粒体DNA多态研究和应用进展. 中国水产科学, 1998, 5(3): 94-103. |
 2015, Vol. 27
2015, Vol. 27 

