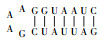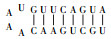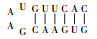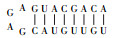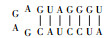(2: 中国科学院藻类生物学重点实验室, 武汉 430072)
(3: 中国科学院大学, 北京 100049)
(2: Key Laboratory of Algal Biology, Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072, P. R. China)
(3: University of Chinese Academy of Sciences, Beijing 100049, P. R. China)
现代生物技术的发展,特别是分子生物学的快速发展,为蓝藻的多样性研究提供了更多的信息和证据.蓝藻的分类系统特别是在属水平上的分类修订也一直在持续不断地进行.在2000—2014年已经成立了66个蓝藻新属[1].在属以上更好的分类阶层和系统也发生了较大的变化,从1970s—1980s的经典四目系统,到2005年Hoffmann等提出的四亚纲七个目系统[2],一直到2014年Komárek等提出的八目系统[1].这些新的分类修订为蓝藻建立更加合理的分类体系做出了较好的尝试,为在更大范围和更重要的类群中分类修订打下了较好的基础.但是蓝藻内部属的自然分类系统的建立,仍是今后蓝藻分类系统和分类修订的基础工作.
双囊藻属(Geminocystis)是2009年Korelusova等从集胞藻属(Synechocystis)分离出来的新属.在对许多集胞藻藻种的DNA检测结果显示,Synechocystis PCC6308的16SrRNA基因序列同集胞藻的模式种类(S. aquatilis)以及Synechocystis PCC6803的16SrRNA基因序列相似度低于90 %,这个数值已经明显地低于细菌分类和现代蓝藻分类的属的范围,所以主要根据Synechocystis PCC6308的这个特征建立了双囊藻以及模式种类海氏双囊藻(Geminocystis herdmanii),现在双囊藻属还有另外一个种类巴布亚双囊藻(Geminocystis papuanica).进一步研究表明,双囊藻在系统进化上是蓝菌藻(Cyanobacterium)的姐妹类群[3].
在我们调查中国的蓝藻多样性特别是以前较少关注的色球藻目的多样性中,我们从青海湖的封闭子湖——月牙湖采集分离到一株蓝藻藻株,通过16S rRNA基因序列的检测,发现此藻株和双囊藻的藻种显示出最高的相似性,并且聚为一束.进一步进行形态学和超微结构的观察研究,确定此藻株为双囊藻的一个种类.这也是我国对双囊藻属的新纪录属的报道.
1 材料与方法 1.1 样品的采集于2017年5月在青海省青海湖南岸二郎剑风景区附近的月牙湖水域(36°39′18.0″N,100°22′14.8″E,海拔3197 m)使用25#浮游生物网采集水体表层浮游藻类,采集地环境因子如下:水体盐度6‰;温度17.1℃;pH值9.05.
1.2 藻种分离使用灭菌过的新鲜CT培养基对25#浮游生物网采集的水样进行富集培养(培养温度为25℃,光照强度为25 μE/(m2 ·s),光暗周期为12 h :12 h)[4],10天后采用经典的毛细管分离法对富集后的培养物进行藻种分离:将巴斯德吸管做成毛细管,在100倍倒置显微镜下挑取单根藻丝或单藻细胞,移取至无菌水中清洗5~6次,然后转移至预装有灭菌过的新鲜CT培养基的24孔细胞培养板中培养(培养方法同上).培养20天后可得到纯藻种,纯化后的藻种转接到含有5 ml CT培养基的螺口玻璃管中,保存在中国科学院水生生物研究所有害藻类生物学学科组的藻种库中,藻种编号为CHAB6541.
1.3 藻细胞形态观察和主要光合色素检验藻体形态的观察使用Nikon eclipse 80i型光学显微镜,外接1270万像素的数码相机(DS-Ri1)和相适应的台式计算机.数码拍照及数据测量通过其附带的图像分析软件NIS-Elements D 3.2进行.在拍照及测量过程中,随机选取50个以上的藻细胞进行各项数据的测量与统计.细胞色素检验使用叶绿素a(激发波长440 nm,发射波长680 nm)、藻红蛋白(激发波长525 nm,发射波长570 nm)和藻蓝蛋白(激发波长595 nm,发射波长630 nm).
1.4 藻细胞DNA提取和16S rRNA基因序列的测定对纯化的藻株扩大培养后,参考Lin等[5]的方法进行DNA的提取.用于扩增16S rDNA及16S~23S间ITS序列的引物为PA和B23S [6-7],由上海英骏生物技术有限公司合成. PCR反应体系为50 μl,包含200 mmol/L dNTP、1.5 mmol/L MgCl2、1×buffer PCR缓冲液、10 pmol引物、1 U Taq DNA聚合酶. PCR反应条件为:94℃预变性5 min;94℃变性30 s,55℃退火30 s,72℃延伸2 min,35个循环;72℃延伸5 min. PCR扩增产物用1 %的琼脂糖凝胶电泳检测,通过胶回收试剂盒回收(Bio Flux),克隆到载体pMD18-T上(Takara, Japan).将克隆载体转入感受态细胞E.coli DH5α中,阳性克隆送武汉艾康健生物科技有限公司进行双向测序,测序后得到序列片段用Bio Edit软件拼接.
1.5 分子系统学分析序列通过NCBI的BLAST进行比对,从GenBank数据库中下载的基因序列用于系统进化树的构建.将所有序列用MAFFT v7.312软件进行多重序列比对后[8],对多重序列比对结果运用Mega v.7.0.14软件做保留保守区,去除高变区及模糊区处理.对获得的多重序列比对结果在Jmodeltest 2软件上采用赤池信息量准则(AIC)标准选择核酸替代模型(GTR+I+G)[9],用于贝叶斯分析(BI)和最大似然法(ML)分析,并再次采用Mega v.7.0.14软件以邻接法(NJ tree)构建系统发育树.邻接法采用Maximum Composite Likelihood为核酸替代模型,步展值(bootstrap value)设为1000,空位或缺失位点均当作配对删除(pairwise deletion)处理.最大似然法分析采用IQ-TREE软件[10],模型为GTR+I+G,步展值设为1000.贝叶斯分析采用MrBayes v3.2.6以随机树起始[11],运行10000000代,每100代取样,剔除25 %老化样本,至平均标准偏差低于0.01以下,根据剩余的样本构建进化树,分枝支持率用贝叶斯后验概率表示. ML、NJ和BI 3个系统发育树均以Gloeobacter violaceus PCC 7421为外类群.
1.6 ITS二级结构分析ITS(转录间隔取)D1-D1’螺旋结构和Box-B螺旋采用Mfold 3.2[12]软件构建ITS二级结构,设置均为默认.
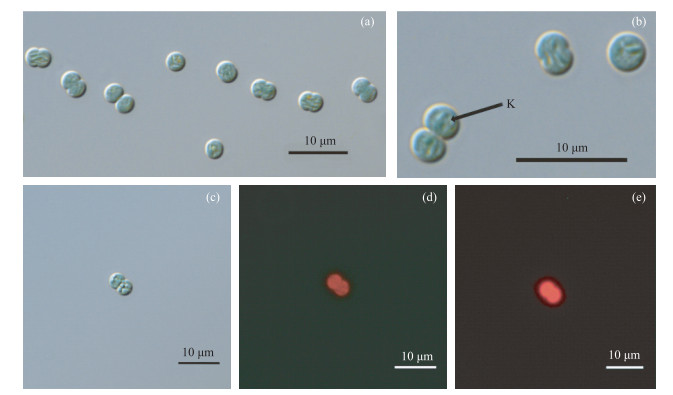
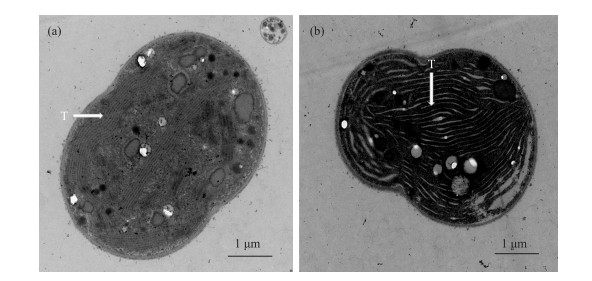
2 结果 2.1 双囊藻的形态描述在液体CT培养基培养状态下,细胞球形、微卵形或微椭圆形,细胞颜色为浅蓝色、亮绿色,衰亡期细胞逐渐呈灰色(图 1a~c).单生,无胶被,不形成菌落.细胞直径范围为2.71~4.22 μm,平均直径为3.34 μm.细胞成分均质,含有叶绿素a(图 1d)和藻蓝素(图 1e),无明显藻红素(结果不显示).细胞纵轴具有窝式结构(keritomized content),为类囊体在细胞纵向的排列(图 1b,图 2).超微结构显示细胞的类囊体排列不规则,有周生加上中生排列,也有很多明显的纵向排列(图 2b).细胞无气囊结构,分裂方式为二分裂.
|
图 1 光学显微镜下Geminocystis sp. CHAB6541的形态(a、b、c为可见光下CHAB6541细胞形态; d为CHAB6541细胞中的叶绿素分布; e为CHAB6541细胞中的藻蓝素分布; K为窝式结构) Fig.1 Light microscopic image of Geminocystis sp. CHAB6541 (a-c show cell morphology of CHAB6541 under visible light; d shows distribution of chlorophyll in CHAB6541 cells; e shows distribution of phycocyanin in CHAB6541 cells; K= keritomized content) |
|
图 2 透射电子显微镜下的Geminocystis sp. CHAB6541的形态 (a和b分别代表正常生长细胞和衰亡期细胞的超微结构, T为类囊体) Fig.2 Transmission electron microscopy(TEM) images of Geminocystis sp. CHAB6541 (a and b show ultrastructure of normal cells and aged cells; T represents thylakoid) |
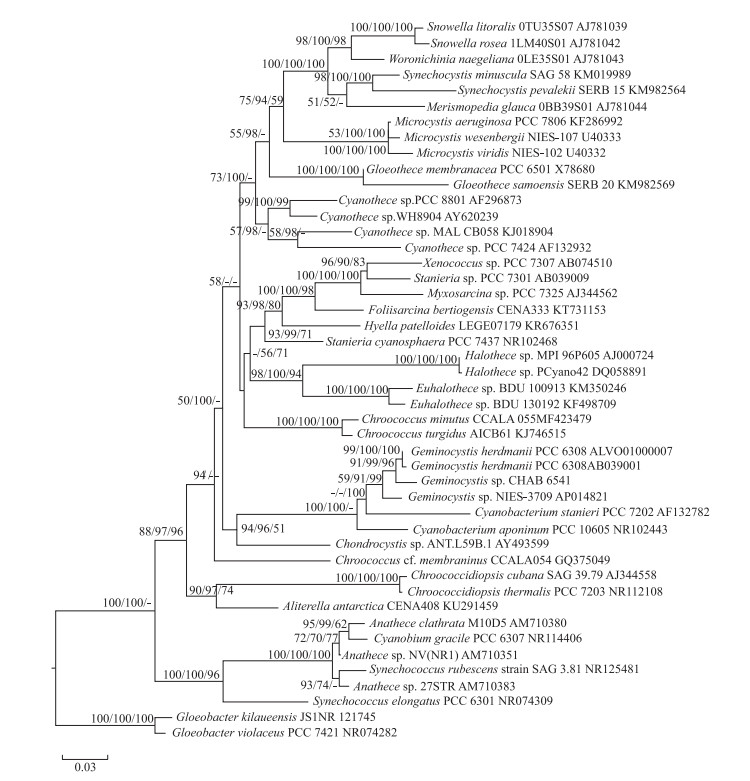
采用上述引物扩增CHAB6541基因组,获得了全长为1862 bp的16S rRNA、ITS和部分23S rRNA序列,Genbank号为MH094654.其16S rRNA序列部分在NCBI进行Blast分析发现,CHAB6541藻株与来自日本的NIES3708和NIES3709及法国巴斯德藻种库的Synechocystis sp. PCC6308表现出较高的相似性(表 1).截取了最大可使用的1305 bp多重比对位点,构建系统发育树,其结果表明,CHAB6541同Geminocystis属下藻株聚类在一起(图 3).
| 表 1 Geminocystis属下不同藻株的16S rRNA基因序列相似性比较 Tab. 1 The 16S rRNA gene evolutionary distances matrix (%) among different strains of Geminocystis |
|
图 3 基于16S rRNA基因序列的ML/BI/NJ分子系统树 (节点处数字表示在ML/BI/NJ中大于50 %的支持数值) Fig.3 ML/BI/NJ phylogenetic trees based on 16S rRNA sequences (Numbers at each node represent over 50 % supporting values in ML/BI/NJ phylogenetic trees) |
CHAB6541的ITS序列上含有Ile和Ala两个tRNA编码基因,其二级结构与Komárek等[1]发表的二级结构略有差异(表 2). Geminocystis sp. CHAB6541和Geminocystis herdmanii PCC 6308的D1-D1’螺旋结构相同:基部茎包含一7 bp螺旋,末端环状结构含有4 bp.两株双囊藻的Box-B螺旋结构不同:Geminocystis sp. CHAB6541基部茎包含一8 bp螺旋,末端环状结构含有4 bp;而Geminocystis herdmanii PCC 6308基部茎包含一6 bp螺旋,虽然末端环状结构同样含有4 bp,但是环状结构5’端一腺嘌呤变为鸟嘌呤.两株双囊藻的V3结构也不同:Geminocystis sp. CHAB6541基部茎包含一8 bp螺旋,末端环状结构含有4 bp;而Geminocystis herdmanii PCC 6308基部茎包含一7 bp螺旋,末端环状结构相同.
| 表 2 Geminocystis sp. CHAB6541和Geminocystis herdmanii PCC 6308的16S~23S rRNA内部转录间隔区(ITS)二级结构的比较 Tab. 2 Comparison of secondary structures of ITS of 16S-23S rRNA between Geminocystis sp. CHAB6541 and Geminocystis herdmanii PCC 6308 |
近年来,我国湖泊生态系统中蓝藻的种类结构和丰度引起了关注,特别是水华蓝藻.所以对蓝藻多样性研究的进展和动态,例如新的类群和记录,也需要密切跟踪.蓝藻的分子系统和分类修订是现代蓝藻多样性研究的主要内容.新属的创建也是蓝藻分类修订的主要部分.本研究中的双囊藻和集胞藻以及蓝菌藻属在最新的Komárek(2014)分类系统中都属于色球藻目的色球藻科、平裂藻科和聚球藻科[1].特别是集胞藻Synechocystis sp. PCC 6803是在1997年蓝藻中的第1个全基因组测序的藻种,也因此成为蓝藻基础生物学研究的最重要和最常用的模式生物.由于单细胞结构的特性,很多有关微囊藻水华生物学特征和过程的研究都是以Synechocystis sp. PCC 6803作为模式生物来进行比较和类推的[3]. Synechocystis sp. PCC 6308非常容易和Synechocystis sp. PCC 6803混淆,一直也是归类在相同的Synechocystis属内.可是由于16S rRNA基因序列的较大差别(小于90 %相似度),将Synechocystis sp. PCC 6308定为蓝藻里的新属——双囊藻属(Geminocystis)[3].根据细菌学分类方法,属和种水平上的分界线分别设立为16S rRNA基因序列的相似度95 %和97 % [1],所以双囊藻属的建立也是得到认可的.通过进一步观察研究,发现双囊藻属的细胞内有条状窝式结构.只有蓝藻里极少数的几个属具有这种窝式结构,例如我们在洱海发现的丝状蓝藻常丝藻属(Tychonema)[13].超微结构也显示双囊藻细胞的类囊体排列不规则.所以,在形态、超微结构以及DNA分子序列上双囊藻都显示出和集胞藻属不同的地方.近年来双囊藻在希腊、巴西和日本等地也相继被报道[14-16],特别是从在日本溪流中分离的Geminocystis sp. NIES3709的全基因组被测序发表[16],显示出对双囊藻属蓝藻的关注度增加.全球已报道的双囊藻属的藻种生境和生态习性显示出非常高的多样性,有湖泊、溪流、土壤、温泉和盐池等.通过检测16S rRNA基因序列,发现从青海湖地区分离的单细胞蓝藻藻种CHAB6541的序列和双囊藻属内的几个藻种序列的相似性最高,都大于97 %,并且在分子系统树中聚为一簇.观察藻种形态和超微结构,发现了细胞内窝式结构和不规则的类囊体排列,进一步证实了我们在属水平上的鉴定结果.在种的鉴定上,ITS二级结构和Geminocystis herdmanii PCC 6308很相近,但是由于和已经报道的藻种的16S rRNA基因序列相似度只有97 %左右(表 1),所以现阶段并不能很好地鉴定到种类,还是以Geminocystis sp. CHAB6541表示.双囊藻属在我国属于首次报道,也显示了我国蓝藻生物的潜在多样性.此次在青海湖地区的小湖泊中发现的浮游习性双囊藻,因此更加关注其是否会在我国更多生境和更多的湖泊中,特别是长江中下游的浅水富营养化湖泊中有分布.我们期待更加先进的生物测定技术如宏基因组和高通量测序技术在湖泊蓝藻研究中的纵深应用能回答这个问题并带来新的蓝藻多样性的信息.
4 结论本研究从我国青海湖地区的小湖泊中分离到一株单细胞蓝藻藻株,通过16S rRNA基因序列显示此藻株和双囊藻的已报道的藻种显示出最高的相似性,都在97 %以上,并且在分子系统树聚为一束.此蓝藻株的细胞结构以及超微结构显示细胞纵轴具有窝式结构以及类囊体不规则的排列,进一步确认它具有双囊藻所拥有的独特特性.这是我国首次报道浮游特性的双囊藻新纪录属.
| [1] |
Komárek J, Kaštovský J, Mareš J et al. Taxonomic classification of cyanoprokaryotes (cyanobacterial genera) 2014, using a polyphasic approach. Preslia, 2014, 86(4): 295-335. |
| [2] |
Hoffmann L, Komárek J, Kaštovský J. System of cyanoprokaryotes (cyanobacteria)-state in 2004. Algological Studies, 2005, 117(1): 95-115. DOI:10.1127/1864-1318/2005/0117-0095 |
| [3] |
Korelusova J, Kaštovský J, Komárek J. Heterogeneity of the cyanobacterial genus Synechocystis and description of a new genus, Geminocystis. Journal of Phycology, 2009, 45(4): 928-937. DOI:10.1111/j.1529-8817.2009.00701.x |
| [4] |
Ichimura T. Isolation and culture methods of algae. Methods in Phycological Studies, 1979, 294-305. |
| [5] |
Lin S, Shen J, Liu Y et al. Molecular evaluation on the distribution, diversity, and toxicity of Microcystis (Cyanobacteria) species from Lake Ulungur-a mesotrophic brackish desert lake in Xinjiang, China. Environmental Monitoring and Assessment, 2011, 175(1/2/3/4): 139-150. |
| [6] |
Edwards U, Rogall T, Blöcker H et al. Isolation and direct complete nucleotide determination of entire genes. Characterization of a gene coding for 16S ribosomal RNA. Nucleic Acids Research, 1989, 17(19): 7843-7853. DOI:10.1093/nar/17.19.7843 |
| [7] |
Lepère C, Wilmotte A, Meyer B. Molecular diversity of Microcystis strains (Cyanophyceae, Chroococcales) based on 16S rDNA sequences. Systematics and Geography of Plants, 2000, 275-283. |
| [8] |
Katoh K, Standley DM. MAFFT multiple sequence alignment software version 7:improvements in performance and usability. Molecular Biology and Evolution, 2013, 30(4): 772-780. DOI:10.1093/molbev/mst010 |
| [9] |
Darriba D, Taboada GL, Doallo R et al. JModelTest 2:more models, new heuristics and parallel computing. Nature Methods, 2012, 9(8): 772. |
| [10] |
Trifinopoulos J, Nguyen LT, von Haeseler A et al. W-IQ-TREE:a fast online phylogenetic tool for maximum likelihood analysis. Nucleic Acids Research, 2016, 44(W1): W232-W235. DOI:10.1093/nar/gkw256 |
| [11] |
Ronquist F, Teslenko M, Van Der Mark P et al. MrBayes 3.2:efficient Bayesian phylogenetic inference and model choice across a large model space. Systematic Biology, 2012, 61(3): 539-542. |
| [12] |
Zuker M. Mfold web server for nucleic acid folding and hybridization prediction. Nucleic Acids Research, 2003, 31(13): 3406-3415. DOI:10.1093/nar/gkg595 |
| [13] |
Wei ZH, Zhu ML, Yu GL et al. Occurrence of planktonic cyanobaterium Tychonema bourrellyi in Erhai Lake and its taxonomic studies. Acta Hydrobiologica Sinica, 2012, 36(6): 1171-1175. [卫志宏, 朱梦灵, 虞功亮等. 洱海的浮游蓝藻布氏常丝藻及其分类学的讨论. 水生生物学报, 2012, 36(6): 1171-1175.] |
| [14] |
Bravakos P, Kotoulas G, Skaraki K et al. A polyphasic taxonomic approach in isolated strains of Cyanobacteria from thermal springs of Greece. Molecular Phylogenetics and Evolution, 2016, 98: 147-160. DOI:10.1016/j.ympev.2016.02.009 |
| [15] |
Andreote APD, Vaz MGMV, Genuário DB et al. Nonheterocytous cyanobacteria from Brazilian saline-alkaline lakes. Journal of Phycology, 2014, 50(4): 675-684. DOI:10.1111/jpy.2014.50.issue-4 |
| [16] |
Hirose Y, Katayama M, Ohtsubo Y et al. Complete genome sequence of cyanobacterium Geminocystis sp. strain NIES-3709, which harbors a phycoerythrin-rich phycobilisome. Genome Announcements, 2015, 3(2): e00385-15. |
 2019, Vol. 31
2019, Vol. 31