eDNA (environmental DNA)是指从环境样品(水体、土壤、沉积物、空气、混合物等)中提取的DNA,是各种生物的DNA混合物,区别于传统从单物种样品中提取的DNA[1-2]。eDNA包括来自活的细胞和生物的胞内DNA,以及来自自然死亡细胞及细胞结构破碎后所释放出来的胞外DNA[1]。
eDNA的应用最早可追溯到1987年对沉积物中微生物的研究[3],但正式兴起则要到2000年之后[1]。应用eDNA的研究早期主要针对微生物类群,扩展到其它生物类群(比如动物)则要略晚几年[4],随着二代测序(next-generation sequencing)和DNA (meta)barcoding技术的发展,生态学中应用eDNA的研究逐渐增多[1, 5],近年来eDNA的应用更是在微生物生态学[6-8]、生态系统生态学[9]、古生态学[10-11]、保护生物学[12]、入侵生物学[13]、特定类群监测[14-15]、生物多样性监测[16-19]等各个研究领域中快速发展[20-21],样品类型覆盖土壤[8, 22]、水体[6, 22]、沉积物[10-11]、气溶胶及碎屑浮尘[23-24]、动物粪便[25-26]、动物肠道[27-30]、动植物体表[27-28, 30]等,目标类群涉及微生物[6-8, 27, 29]、浮游生物[31-32]、真菌[33-34]、植物[35]、低等无脊椎动物[14, 19]、软体动物[36-37]、节肢动物[38]、鱼类[39-41]、两栖爬行类[41-42]、鸟类[23-24]、哺乳类[15, 18]等几乎所有生物类群(表 1)。
| 表 1 eDNA应用概述 Tab. 1 Summary of the eDNA usage in different study fields, samples and taxonomies |
eDNA在各研究领域中的应用,核心一环是利用eDNA对相关生物类群实现信息监测检出及其相对丰度的定量[12-13, 16, 43-44]。eDNA作为标识特定生物存在与否及其相对丰度的指征信号,可以在两个维度发挥功能:1)指征DNA来源物种本身的存在及其相对丰度,2)指征DNA来源物种紧密相关的其它物种或生态环境状况[45]。
2 eDNA监测技术发展的4个阶段eDNA监测技术的发展可划分为技术探索、技术验证、技术标准化、技术常态化应用4个阶段(图 1)。第一次利用沉积物中的eDNA开展微生物研究[3],开启了eDNA监测的技术探索阶段;二代测序和DNA (meta)barcoding技术的成熟实现了eDNA监测技术可用[5],开启了eDNA监测的技术验证阶段,推动了eDNA监测在各领域研究中验证性应用(表 2)[18-19, 23, 43, 46-47];随着eDNA监测可行性在各领域研究中获得验证,同时也认识到eDNA监测结果受各种因素影响[48-50],为了夯实基于eDNA监测的研究结果的有效性,开始研究eDNA监测的技术标准[22, 40, 51]、呼吁eDNA监测的规范化[52-53]、尝试为eDNA监测制定技术标准[54-55],开启了eDNA监测的技术标准化阶段;经过技术发展和知识积累,在eDNA监测完成技术标准化后,eDNA监测将进入诸多学者所预期的技术常态化应用阶段[10, 13, 16-17]。
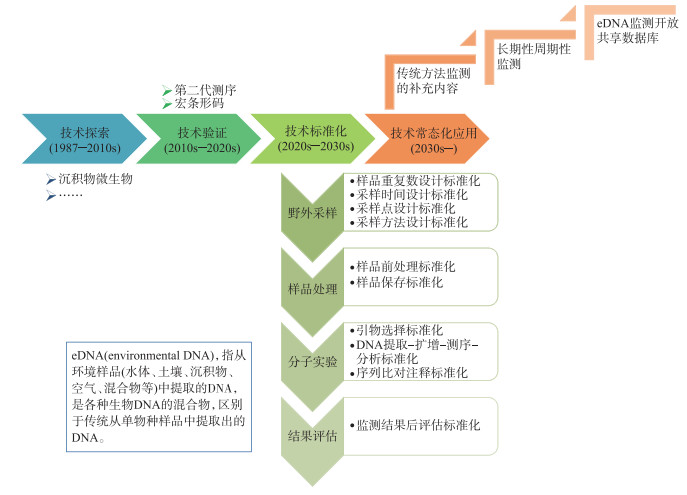
|
图 1 eDNA监测的4个发展阶段 Fig.1 Four development stages of eDNA monitoring |
就当前来看,基于eDNA监测的研究整体上处于技术验证向技术标准化过渡的阶段。当前基于eDNA监测的研究多属于技术验证的研究[18, 56-59],只有少部分可归类为技术标准化和数据积累的研究[22, 40, 60]。鉴于eDNA监测技术可以实现非接触、无损伤、高灵敏度、低人力物力时间成本消耗的多物种(或高级分类单元)监测的应用优势[47, 61-63],可以预见未来数年内,在一些当前大家比较关注的问题上,技术和数据积累可能很快就能完成。近年来,国外[54-55]和国内①陆续有eDNA监测技术标准化文件/手册推出,其已经集中关注了采样点(河流、湖泊、库塘等)选择、样品(水、沉积物、生物膜、混合生物样等)采集与处理、eDNA提取、eDNA分析(扩增、测序、注释、统计)等内容。这些努力推动了eDNA监测技术标准化的进步,但在其所关注的内容中还依然存在不少问题有待解决[51],并且还有一部分问题未受到应有的关注。也就是说,在一些未被关注问题的标准化研究和数据积累上,或有可能掉队,进而延误整个技术标准化的发展。因此,构建一个针对eDNA监测技术标准化的系统性整体框架是非常必要的,将有助于推动eDNA监测技术尽快发展到技术常态化应用阶段。
① http://www.chinacses.org/xw/gsgg/202205/t20220516_982140.shtml.
| 表 2 eDNA监测在各领域研究中的验证性应用 Tab. 2 The test of eDNA monitoring in different study fields |
针对eDNA监测技术标准化的系统性整体框架的需求,本文借鉴文献中所指出的eDNA监测技术中可能面临的一系列问题[12, 17, 48-50, 52],并对eDNA监测技术流程进行系统化梳理(图 2),提出eDNA监测技术标准化中所需重点关注和解决的10个环节(图 1):样品重复数设计、采样时间设计、采样点设计、采样方法设计、样品前处理、样品保存、扩增引物选择、样品提取-扩增-测序-分析程序、序列比对注释、监测结果后评估,并就此进行简要说明和分析。
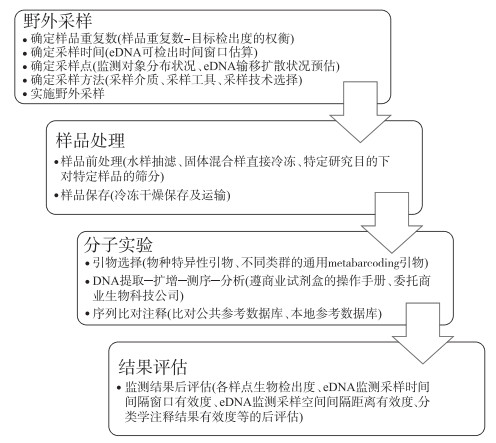
|
图 2 eDNA监测技术流程 Fig.2 Technological process of eDNA monitoring |
该系统性整体框架主要针对基于eDNA metabarcoding技术路径的eDNA监测方法。基于eDNA监测技术发展历程和未来应用图景,我们判断未来eDNA监测技术发展应用将是以基于eDNA metabarcoding的技术路径为主,以非基于eDNA metabarcoding的技术路径为辅,同时考虑到非基于eDNA metabarcoding的技术路径的eDNA监测技术流程比基于eDNA metabarcoding的技术路径的eDNA监测技术流程只在“扩增引物选择、样品提取-扩增-测序-分析程序、序列比对注释”3个环节有差异或省略,所以本文着重阐述基于eDNA metabarcoding技术路径的eDNA监测方法的标准化框架,兼顾非基于eDNA metabarcoding技术路径的eDNA监测方法的标准化框架。
3.1 样品重复数设计eDNA监测样品重复数设计的核心是样品重复数与目标检出度之间关系的测算。eDNA监测属于抽样调查,抽样重复数越多检出的信息种类数也越多(同时,检出的信息相对丰度结构也越稳定),这种增加遵循物种积累曲线的对数增长基本规则,所以会存在一个样品重复数和目标检出度之间的相对优化的投入产出点[40]。比如,在长江武汉江段开展的鱼类eDNA监测中,10个重复可以实现近80% 的鱼类物种检出度目标,13个重复可以实现近90% 的鱼类物种检出度目标,17个重复可以实现近95% 的鱼类物种检出度目标[40]。在长江武汉江段开展的环境微生物eDNA监测中,8个重复可以实现近80% 的细菌物种检出度目标,17个重复可以实现近90% 的细菌物种检出度目标,28个重复可以实现近95% 的细菌物种检出度目标[29]。
样品重复数设计,主要是在一个相对狭小的时间和空间内,通过对采样时间和采样空间的离散设计,以随机抽样假重复的方式采集一系列样品,通过样品的处理、测序、分析、注释、物种积累曲线分析,计算最大可检出物种数(operational taxonomic units,OTUs)的最优估值,获得样品重复数和目标检出度之间的量化关系,然后根据一定的检出度目标,确定eDNA监测所需的样品重复数[40]。针对单物种监测的样品重复数设计,可以根据一系列重复抽样监测中目标物种群体的检出频次,进行基于0/1随机抽样模型的概率计算来确定样品重复数与目标检出度的关系。
样品重复数设计的标准化原理简单,只需要在各具体区域针对各具体目标类群在各具体环境条件范围内开展实验积累数据,建立标准参考库,然后根据特定目标检出度确定样品重复数,或者根据样品重复数与目标检出度关系选取具有最优投入产出的样品重复数。
3.2 采样时间设计eDNA监测采样时间设计的核心是监测时间有效度测算。因为eDNA在环境中存在一定的持留时间[60, 64],所以eDNA监测的采样时间设计需要解决两个问题:1)怎么避免前后两次采样监测到的是同一批信号源,2)怎么避免出现大的监测时域空缺,这需要对eDNA的监测时间有效度进行量化测算。
监测时间有效度,指在监测区域内监测目标群体的eDNA降解至不可检出的时间。不可检出取决于两个层面的因素:eDNA片段降解至短于监测扩增片段长度[65],可检出eDNA片段的浓度降解或稀释至低于检出限[66]。由于监测时间有效度受eDNA初始片段长度、初始浓度、降解速率等的影响[60, 64],eDNA的释放形态和释放率存在较强的类群差异性[67],eDNA降解速率受来源(生物类群)[67]、形式(胞内胞外)[68]、初始浓度[69]、温度[70-72]、赋存介质[73]、BOD[69]、营养盐组成与浓度[68, 70]、pH[72, 74]、紫外线[72]、叶绿素[69]、环境微生物[65]及胞外酶等因素影响[20, 75],监测时间有效度呈现出物种差异性、类群差异性、季节差异性、环境特征差异性等特征[60, 76]。
在陆地上和静水水体中(以及其它的一些类似情况,比如附着于器物表面),采样时间设计主要是在一个具体环境条件下,对实际目标生物类群密度条件下所产生的eDNA的降解过程进行定量。因为eDNA的降解存在指数降解的规律[20, 69-70, 77],所以可以用半衰期来进行定义[65]。因为生物个体所释放的eDNA都是从大片段降解为小片段再降解为更小的片段,因而eDNA长片段的半衰期比短片段的半衰期更短[64-65, 78],用长片段引物比用短片段引物进行eDNA监测时的时间窗口更小。
在流水水体中和强烈受流域过程影响的陆地上(以及其它的一些类似情况,比如受空气流动而传播的气溶胶和碎屑浮尘),采样时间的设计不仅仅需要考虑eDNA降解过程,还要考虑eDNA在水体、沉积物、土壤、空气等采样目标介质中的持留时间。在径流、气流的侵蚀、搬运过程影响下,eDNA会在尚未完全降解的情况下被输移到其它地方[79-82],进而导致原位事实上的eDNA不可检出,因而这种情况下的采样时间设计就要考虑eDNA持留时间这一参数,而非单纯的降解过程。
采样时间设计的标准化影响因素复杂,但针对常规应用的标准化方法简单,只需要在各具体区域针对各具体目标类群在各具体环境条件范围内开展实验积累数据,建立标准参考库,然后根据相应监测时间有效度确定监测采样时间间隔。
3.3 采样点设计eDNA监测采样点设计的核心是监测空间有效度测算。因为eDNA在环境中存在一定的持留时间[60, 64],受径流、气流、扩散等的驱动以及动物携带传播而存在一定的空间迁移[9, 75, 83],所以eDNA监测的采样点设计需要解决两个问题:1)怎么避免两个采样点监测到的是同一个信号源,2)怎么避免出现监测区域大的空间空缺,这需要对eDNA的监测空间有效度进行量化测算。
针对流水系统中的eDNA监测,有学者提出用基于水文学模型,考虑eDNA降解速率以及各环境因子,通过模型模拟计算来设计eDNA监测的最适采样方案[84]。这属于通过受控实验明晰环境因子对eDNA输移、降解过程中各环节的影响[85-86],再通过各环境因子的参数化、eDNA输移降解过程各环境的模型化来实现对eDNA监测的最适采样方案设计的思路[75]。另外一个思路,即暂时先把eDNA产生、输移、降解等的复杂过程(各物种的eDNA形成参数、各物种eDNA的降解速率参数、各环境要素对eDNA降解的影响参数)打包成黑箱,接受不确定性、控制不确定性、量化不确定性,通过对eDNA输入端和输出端关联性的量化分析,实现对eDNA监测的最适采样方案设计,这是我们所提出的流域生物信息流分析框架的处理问题方式[9, 22]。由于该思路所需确定的背景变量少、所需预先开展的计算简单,虽然精确度有所折损,但更适合大范围推广使用。
因为eDNA的降解存在指数降解的规律[20, 69-70, 77],在流水水体中eDNA的监测空间有效度可以用无效生物信息流(用来标记没有生命活性的生物物质,即来自自然死亡细胞及细胞结构破碎后所释放出来的胞外DNA)的半衰距离来量化[22]。因为依赖于eDNA的降解速率以及由径流驱动的运输、吸附与沉积、再悬浮等过程[75, 79-82],其中eDNA的降解速率受来源(生物类群)[67]、形式(胞内胞外)[68]、初始浓度[69]、温度[70-72]、赋存介质[73]、BOD[69]、营养盐组成与浓度[68, 70]、pH[72, 74]、紫外线[72]、叶绿素[69]、环境微生物[65]及胞外酶等因素影响[20, 75],无效生物信息流半衰距离会呈现出物种差异性、类群差异性、季节差异性[22],以及基于河流特征的区域性差异。比如,在青藏高原一个小型河流中,春季封冻期的细菌无效生物信息流半衰距离为1.5 km左右,秋季多云天的细菌无效生物信息流半衰距离为10.5 km左右,夏季降雨天的细菌无效生物信息流半衰距离为14.5 km左右,而夏季降雨天的真核生物无效生物信息流半衰距离为4.5 km左右[22]。因为样品重复数对流域生物信息流的估算准确度和精密度有影响[87],所以采样点设计需要基于具体样品重复数所指向的目标检出度进行,并通过后验概率检验来对eDNA监测空间有效度进行评估。
在静水水体中eDNA的监测空间有效度往往依赖于水体内监测目标类群的空间分布以及eDNA在水体中的扩散方向和范围,监测空间有效度可以用eDNA空间分布差异度和eDNA矢量化有效扩散距离来量化。比如,在不同规模(面积分别为3、122和4343 hm2)的湖泊中的eDNA监测研究显示,近岸水体eDNA样品检出的鱼类物种数比湖中水体eDNA样品检出的鱼类物种数和多样性高,且大多数物种相同[88]。在海洋中的eDNA监测研究显示,受水体垂直分层和水生生物群落结构的垂直分层影响,采样点也需根据需要进行分层设置[89]。
对陆地生物的eDNA监测的采样点设置,可以基于由陆地到水体的流域生物信息流的框架通过河流水体eDNA来监测[9],也可以比照静水水体中eDNA监测框架来开展,只是需要将eDNA在水体中的扩散过程替换成陆地动物移动/携带扩散和风力驱动扩散过程。比如,在青藏高原一个小流域中,离岸5 m内的陆地土壤eDNA中检出的细菌可以在河流水体中检出的比例,在春季封冻期为16.6%,秋季多云天为48.1%,夏季降雨天为62.8%,真核生物的检出普遍要低一些,夏季降雨天的真菌检出比例为44.7%,后生动物的检出比例为22.6% [22]。
对附着于器物或者生物个体(内)表面的eDNA监测的采样点设置,主要依赖于对所监测目标eDNA分布的预判,而这个目标eDNA由监测或研究目的所决定。比如监测动物的肠道微生物,采样点为动物肠道(有的会区分前肠、中肠、后肠)或者其所延伸的排泄物[25, 27-30];监测动物的体表共生微生物,采样点为动物皮肤或者特定器官/区域的外表皮[27-28]。
采样点设计标准化的影响因素复杂,对于存在明确eDNA空间输移的情况,可以选择黑箱处理方法,基于流域生物信息流分析框架,计算eDNA监测的空间有效度,并对其进行概率检验;对于不存在明确eDNA空间输移的情况,可以借鉴生态学中经典的种-面积曲线思路,基于均匀采样设计来量化采样点间距或采样数量与目标检出能力的关系。具体标准化工作只需要在各具体区域针对各具体目标类群在各具体环境条件范围内开展实验积累数据,建立标准参考库,然后根据相应监测空间有效度确定监测样点。
3.4 采样方法设计对不同地区、对象、目的,eDNA监测需要选取不同的样品类型(水体、沉积物、土壤、动物粪便、大型昆虫、物体表面附着物)或样品类型组合。因为生物在分布区域内不同样品介质中的eDNA存留会存在明显差异,不同样品介质对不同的生物eDNA的持留方式、持留能力和持留时间也会存在明显差异,环境条件也会对这两类差异产生显著影响,进而导致不同样品类型所保存的类群不一样,或对各类群的覆盖度不一样,所以在相关监测工作中,要注意选择最适样品类型(组合)进行采样。比如,一个对澳大利亚(印度洋和南大洋)和哈萨克斯坦(里海)港口的eDNA监测研究显示,来自表层水、沉积物、沉降盘、浮游拖网的eDNA样品检出的真核生物类群具有明显分异,不同类型样品eDNA监测到的真核生物类群存在系统性互补[90]。在对西澳大利亚的高温荒漠气候的Pilbara和高温地中海气候的Swan海岸平原的陆生生物eDNA调查研究中发现,食肉动物粪便eDNA样品比土壤eDNA样品能够检出更多的植物和无脊椎动物类群(科级别)[91]。沉积物中的eDNA降解速率比水体中的eDNA降解速率要慢、持留时间要长,同样体积的样品,沉积物中累积的eDNA浓度比水体的要高、时间跨度比水体的要长[73, 92],这可为不同监测目标下的样品类型选择提供参考。因为地上节肢动物留在植物叶面的eDNA容易受雨雾冲刷而下落[93],所以林间节肢动物eDNA监测建议采集植物冠/叶下的露水、雨水或者表层土壤样品。
野外采样量一方面取决于样品类型和eDNA监测目标,另一方面取决于eDNA提取过程中样品的最大使用量。当前eDNA提取最常用的试剂盒法中,eDNA提取所使用的样品量通常不超过1.5 mL。所以对于不需要预处理且能够大量采集的沉积物样品、土壤样品等,可以用5 mL无菌离心管采集3~5 mL样品[22],根据需求可增加样品重复数;对于不需要预处理但样品较不易采集的生物肠道样品、生物体表黏膜样品、物体表面附着物样品、气体悬浮物沉降样品等,可以用2 mL无菌离心管采集0.5~2 mL样品[27, 29],根据需求可增加样品重复数;对于需要通过抽滤来富集eDNA的水体样品,目前多以1.5 L为量(不同研究不同标准对这一量的要求有所差异),在不同水体状况和孔径滤膜条件下,抽滤完所有水样可能需要使用多张(1~5)滤膜,多张滤膜在eDNA提取时可以作为一份样品一次性提取[22, 29],根据需求可增加样品重复数;对于需要通过抽滤来富集eDNA的气溶胶及碎屑浮尘样品,可以基于研究区域的空气悬浮物状况来确定抽滤空气的时间(0.5~2 h)[23-24],根据需求可增加样品重复数。
不同样品(样品重复)不要混样。因为eDNA监测采样的随机抽样特性以及eDNA在介质中非均一分布特性,各样品中所包含的各物种的eDNA丰度不一,丰度超过检出阈值的物种大概率会被检出,低于检出阈值的大概率不会被检出,所以各样品中所检出的群落结构也不一,一个样点所有样品的检出结果(在分析过程中)合并在一起可以更贴近事实地反映其群落结构状况;而不同样品进行混样(但不进行eDNA富集操作)会稀释某个样品所偶然抓住的某低资源量物种超过检出阈值的eDNA丰度,导致一系列低资源量物种的eDNA丰度低于检出阈值,进而导致检出结果的系统性误差。综合分析已有的qPCR实验研究,不同实验室不同研究条件下的eDNA检出限(limits of detection)在4~250个拷贝每个反应(拟合结果范围略有扩张,2.19~260),eDNA量化限(limits of quantification)在10~1920个拷贝每个反应(拟合结果范围略有左偏,6~839)[66]。有案例研究也证实,基于各样点样品单独测序结果的物种累计曲线比基于各样点样品混合样重复测序的物种累计曲线所获得的物种数和多样性要高[88]。
3.5 样品前处理样品前处理主要是指对水样的处理以获得富集的eDNA样品,因为通常情况下土壤、沉积物、固体混合物、痕迹样品采集后直接密封冷冻即可。最高效的水样预处理方式是过滤,不同的过滤方式所得到的结果差异不大,但过滤用的滤膜孔径规格和滤膜材质对结果有明显影响:孔径越小所捕捉到的eDNA类群越多,其代价是过滤时间越长,在实践中需要根据监测目标的形态(病毒、细菌、浮游生物、宏体生物碎片、游离DNA)进行权衡;对于滤膜材质,有研究显示用河流和湖泊水体eDNA监测真核生物,玻璃纤维滤膜比树脂滤膜的过滤效果好,纤维素酯滤膜比聚醚砜滤膜的过滤效果好[94-96]。
不建议对水样进行预先过滤(除了为富集某特定类群eDNA而进行的筛分)。一些研究对较浑浊水样进行预先过滤以去除大颗粒杂质,然后用微孔滤膜过滤获得细颗粒的eDNA样品,但考虑到大颗粒杂质对于用微孔滤膜进行过滤不会产生特别大的影响,以及预处理在除去大颗粒杂质时也会去除相当一部分被大颗粒杂质吸附的目标eDNA,所以对悬浮颗粒较多、泥沙含量较高或富营养化的水体可进行分样处理(通过静置或离心将颗粒物进行富集形成沉淀物样品,按照沉积物样品的处理方式进行eDNA提取,上清液进行抽滤形成滤膜上的样品,进行eDNA提取),也可以延长抽滤时间、压缩抽滤水体体积、增加样品的重复数,但不建议对水样进行预先过滤。一项对海洋浮游动物群落结构的案例研究确证了经过预先过滤的eDNA样品中所检出的生物类群显著少于未经预先过滤的eDNA样品中所检出的[97]。因为预先过滤并不降低样品污染、测序错误的可能性,所以减少的生物类群大概率是稀有物种/类群。eDNA监测的主要目的和优势之一是对稀有物种的检出,而非对传统方法能够轻易得出的结果进行重复验证,所以不进行水样预先过滤的代价是值得的。
对于部分特定目的或针对特定类群的非水体eDNA监测,也可以通过样品前处理来提高监测效率。比如,对在两个鲑鱼养殖场采集的84个底泥样品分组进行筛分对比实验,非筛分样品中后生动物序列(metazoan reads)的平均百分比分别为19.53% 和17.10%;筛分样品中后生动物序列的平均百分比分别为81.03% 和89.92% [98]。筛分有效地清除了硅藻门类群(如海藻和浮游植物),富集了后生动物。筛分样品中eDNA测序中底栖动物平均占比在一个样点是未筛分样品的4.1倍(47.29% vs 11.46%),在另一个样点是未筛分样品的5.7倍(20.03% vs 3.52%)[98]。
3.6 样品保存样品保存主要指样品从被采集到DNA提取之间的这段时间的保存。对于不需要预处理的样品(比如土壤、沉积物、固体混合物、痕迹等)最有效且便捷的方法即-80℃、-20℃或干冰浴冷冻保存。对于水样,冰浴保存可以降低eDNA在水中的降解速率[99],也有用阳离子表面活性剂来抑制水体中eDNA降解[100],但是不如低温保存有效[101]。当然,最有效的还是尽快进行过滤,把eDNA富集在滤膜上,然后干燥冷冻(-80℃、-20℃)保存[96]。
3.7 扩增引物选择扩增引物,根据eDNA监测的对象和目的选择用物种特异性引物或者metabarcoding通用引物。物种特异性引物主要针对特定物种的eDNA(快速)监测检出与定量(比如病源监测、入侵物种监测、濒危物种监测等),metabarcoding通用引物主要针对目标生物类群的eDNA监测检出(比如对细菌、鱼类、植物、真核生物等的物种组成监测)[49]。不同类群往往有不同的常用通用引物(比如真菌常用ITS、18S rRNA基因的通用引物,细菌、古菌常用16S rRNA基因的通用引物,后生动物常用COI、线粒体12S rRNA、Cyt b基因的通用引物)。不同通用引物之间,有一定的扩增类群范围、类群内扩增比例、物种/遗传辨识度差异[102-104]。传统通用引物在越来越多的使用中逐渐形成大势并不断积累数据,同时也有不少新的通用引物被开发出来[35, 105-106],但新开发通用引物是否真的好用,是否有足够的优势替代已塑造了一定程度路径依赖的传统通用引物,还有待实践和时间检验。
对于鱼类,针对线粒体12S rRNA基因的通用引物比针对COI基因的通用引物有更好的检出效果[104, 107]。当前的状况是,针对线粒体12S rRNA基因的通用引物检出结果可重复性高,但数据库不全;针对COI基因的通用引物检出结果可重复性低,但数据库全[107],因而当前用COI的通用引物还是更普遍一些。随着线粒体12S rRNA数据库的完善,线粒体12S rRNA的通用引物之后或将是一个重要的可选项。由于当前各metabarcoding通用引物对目标类群的扩增都存在一定的效率缺陷,使用多个通用引物分别进行扩增测序分析,并对结果进行并联分析能够一定程度上缓解部分覆盖缺陷问题,但是否能够彻底解决覆盖缺陷问题、是否能够准确反映群落结构尚未确定。
对样品进行环境宏基因组(metagenome)测序并系统注释,也是一种检出方式[108]。宏基因组测序能够覆盖各生物类群,所得的数据量大得多,但成本却高得多,结果的精确度和覆盖度也未确定,目前在微生物上用得多,在其它类群中用得非常少,需要根据实际需求进行选择,并对类群覆盖度和注释效率进行相应定量评估。
3.8 样品提取-扩增-测序-分析程序对每一类样品,应该有标准化的eDNA提取-扩增-测序-分析程序。因为eDNA提取方法和具体操作程序、PCR扩增的退火温度和循环数、测序平台和测序技术、分析流程和具体参数都会对最后所解析出来的OTUs丰度评估产生一定影响,所以只有统一标准化的eDNA提取-扩增-测序-分析程序,其最后所得的结果之间才具有完全的可比性[109]。对于具体的eDNA提取方法,有学者提出特定改进方案[110],但用商业试剂盒是越来越普遍的方式[111]。从现状来看,从eDNA商业试剂盒提取、eDNA样品质量检测,到PCR扩增、PCR产物质量检测、PCR产物提取与纯化、PCR产物定量,再到DNA文库构建、测序、序列数据质控、OTU聚类、OTU丰度统计[22],包括其中各个步骤的污染控制、参数设置及调优,商业生物技术公司都有一整套标准化技术流程及数据处理软件,并在为非常多的eDNA相关研究提供技术服务支持。从目前趋势可以预见,样品提取-扩增-测序-分析一整套都委托给商业生物技术公司也将是未来的一个重要选项和趋势[112]。
虽然由商业生物技术公司执行的eDNA样品提取-扩增-测序-分析程序在执行过程中由公司技术人员不断技术参数调优进而逐渐趋于标准化,但还存在两个细节问题尚待解决并值得注意:1)扩增当中的引物偏好问题。该问题的存在比较普遍,并且是对群落结构定量产生重要影响的技术误差来源[113],这个问题的解决一方面需要开发更好的偏好性更小的通用引物来缓解,另一方面也可以通过定量引物偏好,对整体结果进行模拟和逆向校正。2)分析当中的OTU聚类的参数设置问题。该问题是一个比较直观的问题,OTU聚类时序列相似度设置越高,聚类形成的OTU数目就越多,进而可能影响到后续的序列比对和分类学注释。这是一个计算量和结果精确度的权衡,可以在算力允许的情况下自行调试合适的参数,比如97%、99%、99.9%。
对序列的分析,除了OTU聚类分析路径之外,ASV(amplicon sequence variant)降噪分析路径也在逐渐获得相关研究者的关注。从OTU聚类分析路径和ASV降噪分析路径单独使用比较来看,聚类及注释结果各有优缺点,比如针对COI基因所得聚类OTUs和降噪所得ASVs差不多,并且注释所得分类单元也差不多,而针对18S基因所得聚类OTUs明显比降噪所得ASVs要多,并且注释所得分类单元也要多[103]。有研究提出OTU聚类分析路径和ASV降噪分析路径不应该是相互替代的,而是相互补充的,建议用ASV来指示单倍型,用OTU来指示物种,避免相关信息丢失,进而提高不同研究间的可加性和可比性[114]。聚类分析路径和降噪分析路径都有不止一种算法[115-116],相应类别之内的算法在使用中或多或少可互相混合、折中和交换[114],在标准化过程中建议选定一种或几种简易有效的算法进行计算和结果展示。
对于针对特定物种的eDNA(快速)监测检出与定量,往往只需要使用物种特异性引物进行PCR定性检出和qPCR或ddPCR定量分析[49],不需要进行测序、序列分析、序列比对注释等步骤环节。使用物种特异性引物进行qPCR或ddPCR定量分析,主要是进行eDNA相对丰度的定量,因为eDNA相对丰度在一定条件下和目标物种的个体数量呈线性正相关[117-118],进而可被用来开展鱼类自然繁殖监测[59]。通过eDNA定量分析可以为各物种/类群的eDNA释放速率、eDNA降解速率、eDNA浓度与个体数量关系等进行数据积累,进而为未来用模型根据eDNA浓度模拟计算目标类群个体数量或资源量提供数据库支持。
3.9 序列比对注释序列比对注释的关键在于参考数据库的建立与完善。对于特定目标物种的监测(比如特定珍稀濒危物种监测、入侵种监测、寄生虫监测等)只需要通过物种特异性引物进行PCR扩增检测确认即可,无需进行测序和序列比对[13];如果需要对特定目标进行种群遗传结构分析和系统发育分析,则需要进行测序和相关序列分析。对于大类群目标的监测则需要特定的序列参考数据库,比如真菌的ITS基因Unite数据库、18S rRNA基因Silva数据库,细菌和古菌的16S rRNA基因Silva或Greengene数据库,后生动物的线粒体COI基因用NCBI核酸数据库等。对于具体区域的具体类群的eDNA监测往往会因为目标数据的有限性使得常用参考数据库中相关序列存储不全所造成的结果遗漏显得不可接受,所以参考数据库的完善对于eDNA监测的应用非常必要[40]。在公共参考数据库不能满足特定区域特定类群的研究需求时,可以根据已有材料、已知信息建立本地参考数据库[119-120]。
序列比对注释中另一个需要特别注意的问题是分类置信度的设置。在参考数据库中参考序列对物种覆盖不是十分完全的情况下,如果序列比对注释的分类置信度设置得较低,往往会出现一些错误注释,即把某一物种的序列错误匹配到序列相似的另一物种上,或者把缺少参考序列的某一物种的序列强行匹配到序列相似的另一物种上[40],在当前参考数据库建设条件下,后一种情况更为普遍。在参考数据库中参考序列对遗传多样性覆盖不是十分完全的情况下,如果序列比对注释的分类置信度设置得很高,往往会出现一些没有注释的OTU,即该OTU序列通过比对无法与参考数据库中的任一序列形成匹配,这种问题在基于不完备的本地数据库的比对注释中较为常见。这是一个参考数据库完备程度和结果匹配精确度之间的适配,可以自行调试合适的参数,比如97%、99%、99.9%。比对注释所使用的算法差异(比如BLAST、RDP classifier、Bayesian Lowest Common Ancestor)和软件差异(比如Qiime2、Usearch)对结果也会产生一定影响,其结果的准确度需要相应的比较研究来确定,在对不同研究的注释结果进行横向比较时应对算法和软件进行统一。
3.10 监测结果后评估监测结果后评估主要是对监测结果的样品重复数的后评估、时间有效度的后评估、空间有效度的后评估、注释有效度的后评估,是利用当次监测结果和历史参考数据对当次监测过程中相关环节的评估,其它内容不是不需要评估,而只是因为无法根据当次监测结果进行后评估。样品重复数的后评估,即以当次监测所采样品的检出结果分析样品重复数和目标检出度(或特定目标检出概率)之间的关系,计算当次监测所采样品重复数所能达到的检出度水平,是对样品重复数设计的校验。时间有效度的后评估,即以当次监测相邻采样时间各相应采样点所获得的监测结果差异度(组成差异度或丰度差异度),结合可确认的物种差异或群落结构差异,分析eDNA的半衰期,评估当次监测的时间有效度,是对采样时间设计的校验。空间有效度的后评估,即以当次监测各样点所获得的监测结果计算eDNA输移或扩散的半衰距离,评估当次监测的空间有效度,是对采样点设计的校验。注释有效度的后评估,即以当次监测所测定序列注释结果与传统调查结果相比较,以及与其它参考数据库注释结果相比较,评估当次监测中参考数据库注释结果的有效度,是对参考数据库及分类学注释的校验。比如在长江武汉江段开展的鱼类eDNA监测中,用线粒体COI基因宏条形码(引物mlCOIintF/jgHCO2198R)共检出89种鱼类,其中有30种由历史捕捞调查结果所确认,而与之平行开展的捕捞调查监测记录到35种鱼类,其中24种在eDNA监测中被检出[40]。
示例化说明,假如有针对某一小流域的动物多样性监测方案,每个采样点位设计5个样品重复,每2个月开展1次监测,整个样点沿水系及其岸带分布且样点间径流距离10 km,样品采集以溪流水体和岸带土壤为对象,水样通过抽滤获得富集eDNA滤膜,干燥冷冻保存运输,土样直接冷冻保存运输,测序引物选择针对真核生物的COI基因通用引物,样品委托商业生物科技公司进行提取、扩增、测序、分析,将已聚类OTU序列比对公共参考数据库进行分类学注释,获得目标类群基础生物组成及相对丰度信息。样品重复数的后评估,即对每个采样点位5个样品重复的结果进行物种积累曲线分析,估算理论可检出物种数,并根据物种积累曲线模拟获得样品重复数和物种检出度间的量化关系,给出当前监测方案的检出度。时间有效度的后评估,即对每个采样点位的相邻2次监测结果进行纵向比较,分析其相似度(重复度)及其变化规律,获得其动物活动时间差异性和eDNA半衰期,给出当前监测方案的时间有效度。空间有效度的后评估,即对系列采样点每次的监测结果进行横向比较,分析在每次采样时段样点间eDNA输移的半衰距离,给出当前监测方案的空间有效度。注释有效度的后评估,即将分类学注释结果(或结果中某特定类群)与传统分类学调查结果进行对比,评估注释结果的有效度(正确注释的比例、注释结果对传统分类学调查结果的覆盖度等)。
监测结果后评估并不困难,不需要另外投入物力去补充实验,只需要根据已获得结果对相关问题进行后验的分析评估即可。当前的研究绝大多数没有开展监测结果后评估,只是因为没有注意到其必要性。希望这个内容在之后的eDNA监测分析评估工作中能够得到践行。
4 eDNA监测技术应用图景 4.1 未来的eDNA监测应用场景随着生态文明建设的推进,更为系统化精细化的生物监测评估将是未来生态环境适应性管理的基础需求,eDNA监测在未来可凭借其检出率高、所需样品重复数少、所需付出的人力物力时间成本低[121-122]的优势成为传统监测方案的便捷高效的补充方案。比如在加拿大魁北克地区对3种两栖类开展的监测中,用传统主动搜索方法观察到目标物种的溪流中都监测到了相应的eDNA信号,主动搜索没有观察到目标物种的9条溪流中也监测到了相关eDNA信号[121]。在美国新泽西州12个葡萄园的48个地块内开展的入侵生物监测中,eDNA监测的检出率比传统目视查找的检出率高一倍多,要达到95% 的检出度,仅需要开展5个次葡萄园和12个次地块eDNA监测,但对应的需要开展14个次葡萄园和29个次地块独立目视监测[122]。因此,eDNA监测完全可以作为一个补充方案或者延伸方案来应用,以发现那些难以被记录到或者容易被忽略的生物信息。
随着开放科学的推进,更为标准化系列化的生物监测结果将是未来生态环境基础数据透明开放共享的基础需求,对长期定期监测区域或对象的技术标准化eDNA监测作为常规生物监测工具之一,其所获得的标准化系列化监测记录和数据可以为研究者、产业界和政府部门的研究和决策提供通用的科学数据基础(表 3)。研究者、产业界和政府部门可以把eDNA监测纳入其生物监测工具包中,按标准化设计的样点和频度进行长期性周期性监测,生成标准化技术文本和结果文件,形成相关数据的标准化系列化积累,并通过推进数据的通用、集成、开放获取为后续的大尺度、长序列研究及相关决策提供具有重要意义的数据包[123-124]。比如水生动物共生微生物影响着宿主的健康[124-126],同时又受环境微生物的显著影响[29, 127-130],因而对环境微生物进行eDNA监测,不仅可用以评估水体微生态健康状况,也可用以预警水生动物健康状况[29, 124],进而为水生态系统管理提供支持,为水生野生动物保护提供支撑。长序列的eDNA监测结果文件,也可以为全球变化的生态系统响应、生态环境保护的生态恢复效果等的深入研究提供数据源支撑,并为后续的管理决策和应对政策提供科学支持。
| 表 3 未来的eDNA监测应用场景概述* Tab. 3 Summary of the scopes of the eDNA monitoring in future |
未来5~10年或将是eDNA监测技术标准化急速发展的时期,到2030年前后,预期将基本完成采样方案、样品处理方法、分子实验方案、监测结果后评估方法的标准化。届时,热点区域特定研究对象eDNA监测的样品重复数检出度、eDNA的降解时间周期、eDNA空间输移的有效距离、eDNA赋存形态和效率等eDNA生态学过程的关键参数将完成相应积累,eDNA监测方案中的样品重复数设置、时间设置、空间设置、样品介质选择或组合将形成一套比较确定的参数,指导和支撑相关长期定期监测,进而为相关研究提供开放、稳定的系列化数据源。非热点区域的eDNA监测方案中的样品重复数设置、时间设置、空间设置、样品介质选择或组合也可以通过本框架所示的采样方案制定方法而进行确定。
样品处理(样品前处理、样品保存方法)也将通过对各种方法和处理效果的对比,选定可以满足要求的一整套优化处理方法。当前国外[54-55]和国内②已经有相关的标准制定,虽然离完全达成共识成为通用执行标准还有一段距离,但可以预见,在有组织地推动下,有望在领域内达成共识并形成统一规范。分子实验将更多地实现引物选择趋同,室内实验由商业生物科技公司来完成,数据分析依托商业生物科技公司所提供的分析软件分析平台来完成。当前eDNA监测的引物选择已形成部分共识;室内实验大多由商业生物科技公司来完成,而商业生物科技公司所采用的程序基本一致,只是个别细节参数可能需要进一步统一;基础生物信息分析软件的开源获取和广泛使用已实现了数据分析基础构架的初步标准化,只是在结果的展示上需要达成统一,即在结果的展示中哪些结果必须提供。
② http://www.chinacses.org/xw/gsgg/202205/t20220516_982140.shtml.
监测结果后评估(包括对样品重复数、样点空间布设、采样时间设定、注释有效度等的评估)也将随着研究者们逐渐意识到其必要性和重要性,而获得方法标准化上的共识,并随着相关通用计算方法、计算代码、计算程序的发布而得以实现。已有组织提出对研究团队eDNA监测能力进行评估,以适应未来常态化监测[131],当前的标准化整体框架或可为研究团队eDNA监测能力建设提供指引。
更进一步来讲,随着社会需求扩张和科学技术发展之间的循环联动,eDNA监测必然向更快速、便捷、全面、准确、智能、便宜、透明、可得等方向发展,由于eDNA的生态学特性,样品重复数、样点空间布设、采样时间设定等所依赖的科学基础不会发生明显改变,所以eDNA监测未来进一步发展的压力基本在技术进步和管理进步上。技术进步,主要是针对固定监测站点的在线自动化实时监测实时分析实时上报、针对非固定站点的流动便携监测设备的一体化采样分析和数据结果上传以及监测结果的提质和监测成本的压缩。管理进步,主要是多部门多数据源(比如水文数据、水环境数据、气象数据、人类活动数据、生物数据等)的实时集总实时关联分析实时预警,以及周期性数据包的发布等。
4.3 eDNA监测技术亟需解决的几个参数和问题就当前的eDNA监测技术应用来看,有几个技术参数和问题亟需解决,因为这几个技术参数和问题在理论上并不存在困难,只是需要一批相对细致的实验研究来进行数据积累,所以比较容易解决。1)eDNA监测需要设置的样品重复数,即需要尽快积累各个监测区域/监测点不同环境条件下的样品重复数-检出度曲线,给出合适的检出度目标下所需的样品重复数。2)eDNA监测两次采样间的时间间隔,即尽快积累各个监测区域各个季节各个目标生物类群的eDNA半衰时间,给出合适的监测辨别度目标下最小的时间间隔。3)eDNA监测样点的间距设置,即需要对于静水水体和陆地(以及其它无明确eDNA输移的对象),尽快积累各监测区域监测点不同环境条件下的样点-物种差异度曲线,对于流水水体(以及其它存在明确eDNA输移的对象,比如气溶胶和碎屑浮尘),尽快积累各监测河段监测点不同环境条件下的eDNA随径流(气流)输移的半衰距离,给出合适的监测精度目标下所需的样点间距。4)eDNA监测的扩增引物和参考数据库,即尽快完善各个类群的公共参考数据库建设,在公共参考数据库短时间内无法完成的情况下,尽快建立各自的本地参考数据库,并针对各特定目标类群和参考数据库确定合适的OTU聚类序列相似度值和序列比对注释分类置信度。5)eDNA监测结果的定量模型,因为有各种因素引起结果系统性偏移,所以在完全弄清楚各个影响因素和影响过程之前,针对生物类群的eDNA监测,尽快积累各个类群实际群落结构和eDNA监测结果的拟合关系,建立通过eDNA监测结果获得实际群落结构信息的映射模型;针对具体目标物种的eDNA监测,尽快积累种群资源数量/重量和eDNA监测检出概率的量化关系,建立通过eDNA监测检出概率反推种群资源数量/重量信息的映射模型。
4.4 eDNA监测技术标准化系统性整体框架的说明考虑到无法对多种多样的eDNA监测相关工作给出一个确切的标准化方案,也无意对每种eDNA监测工作分别给出一个确切的标准化方案,所以本文未对具体的标准化参数进行综述和论证,只集中于eDNA监测技术标准化系统性整体框架的构建和说明。本文所提出的eDNA监测技术标准化系统性整体框架(图 1,表 4)旨在为eDNA监测技术标准化参与者及eDNA监测技术常态化应用者提供一个用于设计、完善、评估eDNA监测的稳健框架,以促进eDNA监测在特定物种预警监测、特定类群生物多样性监测评估、生态系统健康监测评价等工作中的常规应用,从而获得先前所难以获得的对特定物种及早检出、对生态环境状况深入理解。期待本系统性整体框架能成为一种便利的指引,参与者、使用者通过参与、应用、反馈、修订不断加强和改进eDNA监测技术的有效性、适应性。
| 表 4 eDNA监测技术标准化系统性整体框架的适用概述* Tab. 4 Summary of the eDNA monitoring standardizing framework suitability in different sample types |
目前,eDNA监测技术标准化10个环节中的部分技术标准化问题已在一定程度上得到解决,剩下的问题在具体的目标指引下,也有望在短期内获得一定程度的解决。在当前技术快速进步知识快速积累的时代,只要是通过技术进步和知识积累能够解决的问题,就一定能够解决,技术路径就一定能够走通。期待未来5~10年内,eDNA监测在一些热点区域(比如长江)成为一个长期性周期性的常规监测内容,并建立严谨、透明、开放、可用的高质量eDNA监测系列数据集,推动生物多样性(及隐藏生物多样性(hidden biodiversity))研究的开放参与[132],为第四范式的(生态学)科学研究提供数据驱动支撑[133],为生态系统功能及健康管理的系统设计和快速反应提供深度理解和及时预警。
| [1] |
Taberlet P, Coissac E, Hajibabaei M et al. Environmental DNA. Molecular Ecology, 2012, 21(8): 1789-1793. DOI:10.1111/j.1365-294x.2012.05542.x |
| [2] |
Pawlowski J, Apothéloz-Perret-Gentil L, Altermatt F. Environmental DNA: What's behind the term? Clarifying the terminology and recommendations for its future use in biomonitoring. Molecular Ecology, 2020, 29(22): 4258-4264. DOI:10.1111/mec.15643 |
| [3] |
Ogram A, Sayler GS, Barkay T. The extraction and purification of microbial DNA from sediments. Journal of Microbiological Methods, 1987, 7(2/3): 57-66. DOI:10.1016/0167-7012(87)90025-X |
| [4] |
Ficetola GF, Miaud C, Pompanon F et al. Species detection using environmental DNA from water samples. Biology Letters, 2008, 4(4): 423-425. DOI:10.1098/rsbl.2008.0118 |
| [5] |
Shokralla S, Spall JL, Gibson JF et al. Next-generation sequencing technologies for environmental DNA research. Molecular Ecology, 2012, 21(8): 1794-1805. DOI:10.1111/j.1365-294X.2012.05538.x |
| [6] |
Ren Z, Wang F, Qu XD et al. Taxonomic and functional differences between microbial communities in Qinghai Lake and its input streams. Frontiers in Microbiology, 2017, 8: 2319. DOI:10.3389/fmicb.2017.02319 |
| [7] |
Gusareva ES, Acerbi E, Lau KJX et al. Microbial communities in the tropical air ecosystem follow a precise diel cycle. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(46): 23299-23308. DOI:10.1073/pnas.1908493116 |
| [8] |
Landesman WJ, Freedman ZB, Nelson DM. Seasonal, sub-seasonal and diurnal variation of soil bacterial community composition in a temperate deciduous forest. FEMS Microbiology Ecology, 2019, 95(2): fiz002. DOI:10.1093/femsec/fiz002 |
| [9] |
Yang HL, Du H, Qi HF et al. Watershed biological information flow driven by natural runoff in Shaliu River Basin on Qinghai-Tibet Plateau indicated by environmental microbes. Acta Ecologica Sinica, 2021, 41(9): 3475-3487. [杨海乐, 杜浩, 祁洪芳等. 青藏高原沙柳河流域自然径流驱动的流域生物信息流量化特征——以环境微生物为指标. 生态学报, 2021, 41(9): 3475-3487.] |
| [10] |
Bálint M, Pfenninger M, Grossart HP et al. Environmental DNA time series in ecology. Trends in Ecology & Evolution, 2018, 33(12): 945-957. DOI:10.1016/j.tree.2018.09.003 |
| [11] |
Korosi JB, Thienpont JR, Smol JP et al. Paleo-ecotoxicology: What can lake sediments tell us about ecosystem responses to environmental pollutants?. Environmental Science & Technology, 2017, 51(17): 9446-9457. DOI:10.1021/acs.est.7b02375 |
| [12] |
Thomsen PF, Willerslev E. Environmental DNA—An emerging tool in conservation for monitoring past and present biodiversity. Biological Conservation, 2015, 183: 4-18. DOI:10.1016/j.biocon.2014.11.019 |
| [13] |
Larson ER, Graham BM, Achury R et al. From eDNA to citizen science: emerging tools for the early detection of invasive species. Frontiers in Ecology and the Environment, 2020, 18(4): 194-202. DOI:10.1002/fee.2162 |
| [14] |
Sengupta ME, Hellström M, Kariuki HC et al. Environmental DNA for improved detection and environmental surveillance of schistosomiasis. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(18): 8931-8940. DOI:10.1073/pnas.1815046116 |
| [15] |
Boussarie G, Bakker J, Wangensteen OS et al. Environmental DNA illuminates the dark diversity of sharks. Science Advances, 2018, 4(5): eaap9661. DOI:10.1126/sciadv.aap9661 |
| [16] |
Coble AA, Flinders CA, Homyack JA et al. eDNA as a tool for identifying freshwater species in sustainable forestry: A critical review and potential future applications. Science of the Total Environment, 2019, 649: 1157-1170. DOI:10.1016/j.scitotenv.2018.08.370 |
| [17] |
Deiner K, Bik HM, Mächler E et al. Environmental DNA metabarcoding: Transforming how we survey animal and plant communities. Molecular Ecology, 2017, 26(21): 5872-5895. DOI:10.1111/mec.14350 |
| [18] |
Sales NG, McKenzie MB, Drake J et al. Fishing for mammals: Landscape-level monitoring of terrestrial and semi-aquatic communities using eDNA from riverine systems. Journal of Applied Ecology, 2020, 57(4): 707-716. DOI:10.1111/1365-2664.13592 |
| [19] |
Carraro L, Hartikainen H, Jokela J et al. Estimating species distribution and abundance in river networks using environmental DNA. PNAS, 2018, 115(46): 11724-11729. DOI:10.1073/pnas.1813843115 |
| [20] |
Seymour M. Rapid progression and future of environmental DNA research. Communications Biology, 2019, 2: 80. DOI:10.1038/s42003-019-0330-9 |
| [21] |
Veilleux HD, Misutka MD, Glover CN. Environmental DNA and environmental RNA: Current and prospective applications for biological monitoring. Science of the Total Environment, 2021, 782: 146891. DOI:10.1016/j.scitotenv.2021.146891 |
| [22] |
Yang HL, Du H, Qi HF et al. Effectiveness assessment of using riverine water eDNA to simultaneously monitor the riverine and riparian biodiversity information. Scientific Reports, 2021, 11(1): 1-11. DOI:10.1038/s41598-021-03733-7 |
| [23] |
Lynggaard C, Bertelsen MF, Jensen CV et al. Airborne environmental DNA for terrestrial vertebrate community monitoring. Current Biology, 2022, 32(3): 701-707. DOI:10.1016/j.cub.2021.12.014 |
| [24] |
Clare EL, Economou CK, Bennett FJ et al. Measuring biodiversity from DNA in the air. Current Biology, 2022, 32(3): 693-700. DOI:10.1016/j.cub.2021.11.064 |
| [25] |
Shao XN, Song DZ, Huang QW et al. Fast surveys and molecular diet analysis of carnivores based on fecal DNA and metabarcoding. Biodiversity Science, 2019, 27(5): 543-556. [邵昕宁, 宋大昭, 黄巧雯等. 基于粪便DNA及宏条形码技术的食肉动物快速调查及食性分析. 生物多样性, 2019, 27(5): 543-556. DOI:10.17520/biods.2018214] |
| [26] |
Schattanek P, Riccabona SA, Rennstam Rubbmark O et al. Detection of prey DNA in bat feces: Effects of time since feeding, meal size, and prey identity. Environmental DNA, 2021, 3(5): 959-969. DOI:10.1002/edn3.205 |
| [27] |
Reinhart EM, Korry BJ, Rowan-Nash AD et al. Defining the distinct skin and gut microbiomes of the northern Pike (Esox lucius). Frontiers in Microbiology, 2019, 10: 2118. DOI:10.3389/fmicb.2019.02118 |
| [28] |
Guivier E, Pech N, Chappaz R et al. Microbiota associated with the skin, gills, and gut of the fish Parachondrostoma toxostoma from the Rhône Basin. Freshwater Biology, 2020, 65(3): 446-459. DOI:10.1111/fwb.13437 |
| [29] |
Yang HL, Wu JM, Du H et al. Quantifying the colonization of environmental microbes in the fish gut: A case study of wild fish populations in the Yangtze River. Frontiers in Microbiology, 2022, 12: 828409. DOI:10.3389/fmicb.2021.828409 |
| [30] |
Tong Q, Hu ZF, Du XP et al. Effects of seasonal hibernation on the similarities between the skin Microbiota and gut Microbiota of an amphibian (Rana dybowskii). Microbial Ecology, 2020, 79(4): 898-909. DOI:10.1007/s00248-019-01466-9 |
| [31] |
Hu YX, Peng Y, Li RW et al. Plankton diversity and community characteristics in Danjiangkou Reservoir based on environmental DNA metabarcoding. J Lake Sci, 2021, 33(6): 1650-1659. [胡愈炘, 彭玉, 李瑞雯等. 基于环境DNA宏条形码的丹江口水库浮游生物多样性及群落特征. 湖泊科学, 2021, 33(6): 1650-1659. DOI:10.18307/2021.0604] |
| [32] |
Sun JY, Yang JH, Zhang XW. Identification and biomass monitoring of zooplankton Cladocera species with eDNA metabarcoding technology. Asian Journal of Ecotoxicology, 2018, 13(5): 76-86. [孙晶莹, 杨江华, 张效伟. 环境DNA(eDNA)宏条形码技术对枝角类浮游动物物种鉴定及其生物量监测研究. 生态毒理学报, 2018, 13(5): 76-86. DOI:10.7524/AJE.1673-5897.20180108001] |
| [33] |
Matsuoka S, Sugiyama Y, Sato H et al. Spatial structure of fungal DNA assemblages revealed with eDNA metabarcoding in a forest river network in western Japan. Metabarcoding and Metagenomics, 2019, 3: e36335. DOI:10.3897/mbmg.3.36335 |
| [34] |
Heeger F, Wurzbacher C, Bourne EC et al. Combining the 5.8S and ITS2 to improve classification of fungi. Methods in Ecology and Evolution, 2019, 10(10): 1702-1711. DOI:10.1111/2041-210x.13266 |
| [35] |
Coghlan SA, Shafer ABA, Freeland JR. Development of an environmental DNA metabarcoding assay for aquatic vascular plant communities. Environmental DNA, 2021, 3(2): 372-387. DOI:10.1002/edn3.120 |
| [36] |
Stoeckle BC, Kuehn R, Geist J. Environmental DNA as a monitoring tool for the endangered freshwater pearl mussel (Margaritifera margaritifera L.): A substitute for classical monitoring approaches?. Aquatic Conservation: Marine and Freshwater Ecosystems, 2016, 26(6): 1120-1129. DOI:10.1002/aqc.2611 |
| [37] |
Sansom BJ, Sassoubre LM. Environmental DNA (eDNA) shedding and decay rates to model freshwater mussel eDNA transport in a river. Environmental Science & Technology, 2017, 51(24): 14244-14253. DOI:10.1021/acs.est.7b05199 |
| [38] |
Carraro L, Mächler E, Wüthrich R et al. Environmental DNA allows upscaling spatial patterns of biodiversity in freshwater ecosystems. Nature Communications, 2020, 11: 3585. DOI:10.1038/s41467-020-17337-8 |
| [39] |
Afzali SF, Bourdages H, Laporte M et al. Comparing environmental metabarcoding and trawling survey of demersal fish communities in the Gulf of St. Lawrence, Canada. Environmental DNA, 2021, 3(1): 22-42. DOI:10.1002/edn3.111 |
| [40] |
Yang HL, Wu JM, Zhang H et al. Environmental DNA metabarcoding utilization efficiency in monitoring large river fish species composition: A case study in the Wuhan transect of the Yangtze River. Journal of Fishery Sciences of China, 2021, 28(6): 796-807. [杨海乐, 吴金明, 张辉等. 大型河流中鱼类组成的eDNA监测效率: 以长江武汉江段为例. 中国水产科学, 2021, 28(6): 796-807. DOI:10.12264/JFSC2021-0556] |
| [41] |
Valentini A, Taberlet P, Miaud C et al. Next-generation monitoring of aquatic biodiversity using environmental DNA metabarcoding. Molecular Ecology, 2016, 25(4): 929-942. DOI:10.1111/mec.13428 |
| [42] |
Evans NT, Olds BP, Renshaw MA et al. Quantification of mesocosm fish and amphibian species diversity via environmental DNA metabarcoding. Molecular Ecology Resources, 2016, 16(1): 29-41. DOI:10.1111/1755-0998.12433 |
| [43] |
Ji YQ, Ashton L, Pedley SM et al. Reliable, verifiable and efficient monitoring of biodiversity via metabarcoding. Ecology Letters, 2013, 16(10): 1245-1257. DOI:10.1111/ele.12162 |
| [44] |
Bohmann K, Evans A, Gilbert MTP et al. Environmental DNA for wildlife biology and biodiversity monitoring. Trends in Ecology & Evolution, 2014, 29(6): 358-367. DOI:10.1016/j.tree.2014.04.003 |
| [45] |
Zhang MZ, Sun QY, Chen PX et al. How microorganisms tell the truth of potentially toxic elements pollution in environment. Journal of Hazardous Materials, 2022, 431: 128456. DOI:10.1016/j.jhazmat.2022.128456 |
| [46] |
Bass D, Stentiford GD, Littlewood DTJ et al. Diverse applications of environmental DNA methods in parasitology. Trends in Parasitology, 2015, 31(10): 499-513. DOI:10.1016/j.pt.2015.06.013 |
| [47] |
Deiner K, Fronhofer EA, Mächler E et al. Environmental DNA reveals that rivers are conveyer belts of biodiversity information. Nature Communications, 2016, 7: 12544. DOI:10.1038/ncomms12544 |
| [48] |
Cristescu ME, Hebert PDN. Uses and misuses of environmental DNA in biodiversity science and conservation. Annual Review of Ecology, Evolution, and Systematics, 2018, 49: 209-230. DOI:10.1146/annurev-ecolsys-110617-062306 |
| [49] |
Beng KC, Corlett RT. Applications of environmental DNA (eDNA) in ecology and conservation: Opportunities, challenges and prospects. Biodiversity and Conservation, 2020, 29(7): 2089-2121. DOI:10.1007/s10531-020-01980-0 |
| [50] |
Darling JA, Pochon X, Abbott CL et al. The risks of using molecular biodiversity data for incidental detection of species of concern. Diversity & Distributions, 2020, 26(9): 1116-1121. DOI:10.1111/ddi.13108 |
| [51] |
Loeza-Quintana T, Abbott CL, Heath DD et al. Pathway to Increase Standards and Competency of eDNA Surveys (PISCeS)—Advancing collaboration and standardization efforts in the field of eDNA. Environmental DNA, 2020, 2(3): 255-260. DOI:10.1002/edn3.112 |
| [52] |
Nicholson A, McIsaac D, MacDonald C et al. An analysis of metadata reporting in freshwater environmental DNA research calls for the development of best practice guidelines. Environmental DNA, 2020, 2(3): 343-349. DOI:10.1002/edn3.81 |
| [53] |
Dickie IA, Boyer S, Buckley HL et al. Towards robust and repeatable sampling methods in eDNA-based studies. Molecular Ecology Resources, 2018, 18(5): 940-952. DOI:10.1111/1755-0998.12907 |
| [54] |
Minamoto T, Miya M, Sado T et al. An illustrated manual for environmental DNA research: Water sampling guidelines and experimental protocols. Environmental DNA, 2021, 3(1): 8-13. DOI:10.1002/edn3.121 |
| [55] |
Leese F, Bouchez A, Abarenkov K et al. Why we need sustainable networks bridging countries, disciplines, cultures and generations for aquatic biomonitoring 2.0: A perspective derived from the DNAqua-net COST action. Advances in Ecological Research, 2018, 58: 63-99. DOI:10.1016/bs.aecr.2018.01.001 |
| [56] |
Harper LR, Lawson Handley L, Carpenter AI et al. Environmental DNA (eDNA) metabarcoding of pond water as a tool to survey conservation and management priority mammals. Biological Conservation, 2019, 238: 108225. DOI:10.1016/j.biocon.2019.108225 |
| [57] |
Ling JZ, Jiang YZ, Sun P et al. Application and evaluation of enviro nmental DNA technology in fish diversity research in Xiangshan Bay. Journal of Fishery Sciences of China, 2021, 28(2): 205-214. [凌建忠, 姜亚洲, 孙鹏等. 环境DNA技术在象山港水域鱼类多样性调查中的应用与评估. 中国水产科学, 2021, 28(2): 205-214. DOI:10.12264/JFSC2020-0219] |
| [58] |
Chen JQ, Dong L, Ma XM et al. Microbial community monitoring in Baiyangdian Lake based on eDNA technology. Journal of Agro-Environment Science, 2021, 40(8): 1773-1786. [陈家琪, 董丽, 麻晓梅等. 基于eDNA技术的白洋淀微型生物群落监测. 农业环境科学学报, 2021, 40(8): 1773-1786.] |
| [59] |
Li S, Liu X, Jiang W et al. Application of environmental DNA technology in natural reproduction of the four major Chinese carps in Yichang section of Yangtze River, China. Chinese Journal of Applied Ecology, 2021, 32(6): 2241-2248. [李莎, 刘雪清, 姜伟等. 环境DNA技术在宜昌江段四大家鱼自然繁殖中的应用. 应用生态学报, 2021, 32(6): 2241-2248. DOI:10.13287/j.1001-9332.202106.037] |
| [60] |
Li M, Shan XJ, Wang WJ et al. Studying the retention time of Fenneropenaeus chinensis eDNA in water. Progress in Fishery Sciences, 2020, 41(1): 51-57. [李苗, 单秀娟, 王伟继等. 环境DNA在水体中存留时间的检测研究——以中国对虾为例. 渔业科学进展, 2020, 41(1): 51-57. DOI:10.19663/j.issn2095-9869.20180906005] |
| [61] |
Stat M, Huggett MJ, Bernasconi R et al. Ecosystem biomonitoring with eDNA: Metabarcoding across the tree of life in a tropical marine environment. Scientific Reports, 2017, 7(1): 1-11. DOI:10.1038/s41598-017-12501-5 |
| [62] |
Li HX, Huang XN, Li SG et al. Environmental DNA (eDNA)-metabarcoding-based early monitoring and warning for invasive species in aquatic ecosystems. Biodiversity Science, 2019, 27(5): 491-504. [李晗溪, 黄雪娜, 李世国等. 基于环境DNA-宏条形码技术的水生生态系统入侵生物的早期监测与预警. 生物多样性, 2019, 27(5): 491-504. DOI:10.17520/biods.2018233] |
| [63] |
Li M, Wei TT, Shi BY et al. Biodiversity monitoring of freshwater benthic macroinvertebrates using environmental DNA. Biodiversity Science, 2019, 27(5): 480-490. [李萌, 尉婷婷, 史博洋等. 环境DNA技术在淡水底栖大型无脊椎动物多样性监测中的应用. 生物多样性, 2019, 27(5): 480-490. DOI:10.17520/biods.2018227] |
| [64] |
Jo T, Arimoto M, Murakami H et al. Particle size distribution of environmental DNA from the nuclei of marine fish. Environmental Science & Technology, 2019, 53(16): 9947-9956. DOI:10.1021/acs.est.9b02833 |
| [65] |
Wei N, Nakajima F, Tobino T. A microcosm study of surface sediment environmental DNA: Decay observation, abundance estimation, and fragment length comparison. Environmental Science & Technology, 2018, 52(21): 12428-12435. DOI:10.1021/acs.est.8b04956 |
| [66] |
Klymus KE, Merkes CM, Allison MJ et al. Reporting the limits of detection and quantification for environmental DNA assays. Environmental DNA, 2020, 2(3): 271-282. DOI:10.1002/edn3.29 |
| [67] |
Andruszkiewicz Allan E, Zhang WG, Lavery A et al. Environmental DNA shedding and decay rates from diverse animal forms and thermal regimes. Environmental DNA, 2021, 3(2): 492-514. DOI:10.1002/edn3.141 |
| [68] |
Bochove K, Bakker FT, Beentjes KK et al. Organic matter reduces the amount of detectable environmental DNA in freshwater. Ecology and Evolution, 2020, 10(8): 3647-3654. DOI:10.1002/ece3.6123 |
| [69] |
Barnes MA, Turner CR, Jerde CL et al. Environmental conditions influence eDNA persistence in aquatic systems. Environmental Science & Technology, 2014, 48(3): 1819-1827. DOI:10.1021/es404734p |
| [70] |
Eichmiller JJ, Best SE, Sorensen PW. Effects of temperature and trophic state on degradation of environmental DNA in lake water. Environmental Science & Technology, 2016, 50(4): 1859-1867. DOI:10.1021/acs.est.5b05672 |
| [71] |
Jo T, Arimoto M, Murakami H et al. Estimating shedding and decay rates of environmental nuclear DNA with relation to water temperature and biomass. Environmental DNA, 2020, 2(2): 140-151. DOI:10.1002/edn3.51 |
| [72] |
Strickler KM, Fremier AK, Goldberg CS. Quantifying effects of UV-B, temperature, and pH on eDNA degradation in aquatic microcosms. Biological Conservation, 2015, 183: 85-92. DOI:10.1016/j.biocon.2014.11.038 |
| [73] |
Turner CR, Uy KL, Everhart RC. Fish environmental DNA is more concentrated in aquatic sediments than surface water. Biological Conservation, 2015, 183: 93-102. DOI:10.1016/j.biocon.2014.11.017 |
| [74] |
Seymour M, Durance I, Cosby BJ et al. Acidity promotes degradation of multi-species environmental DNA in lotic mesocosms. Communications Biology, 2018, 1: 4. DOI:10.1038/s42003-017-0005-3 |
| [75] |
Zhang N, Chou BF, Xie YH et al. Research progress of environmental DNA shedding, decay and transport in aquatic ecosystem. Transactions of Oceanology and Limnology, 2021, 43(3): 129-135. [张娜, 仇保丰, 谢艳辉等. 水环境生态系统中环境DNA脱落、衰变和运输过程的研究进展. 海洋湖沼通报, 2021, 43(3): 129-135.] |
| [76] |
Gordon M, van Norman K. Mycelial DNA persistence in a forest soil. Environmental DNA, 2021, 3(6): 1208-1213. DOI:10.1002/edn3.242 |
| [77] |
Saito T, Doi H. Degradation modeling of water environmental DNA: Experiments on multiple DNA sources in pond and seawater. Environmental DNA, 2021, 3(4): 850-860. DOI:10.1002/edn3.192 |
| [78] |
Jo T, Murakami H, Masuda R et al. Rapid degradation of longer DNA fragments enables the improved estimation of distribution and biomass using environmental DNA. Molecular Ecology Resources, 2017, 17(6): e25-e33. DOI:10.1111/1755-0998.12685 |
| [79] |
Barnes MA, Turner CR. The ecology of environmental DNA and implications for conservation genetics. Conservation Genetics, 2016, 17(1): 1-17. DOI:10.1007/s10592-015-0775-4 |
| [80] |
Jerde CL, Olds BP, Shogren AJ et al. Influence of stream bottom substrate on retention and transport of vertebrate environmental DNA. Environmental Science & Technology, 2016, 50(16): 8770-8779. DOI:10.1021/acs.est.6b01761 |
| [81] |
Shogren AJ, Tank JL, Andruszkiewicz E et al. Controls on eDNA movement in streams: Transport, retention, and resuspension. Scientific Reports, 2017, 7: 5065. DOI:10.1038/s41598-017-05223-1 |
| [82] |
Fremier AK, Strickler KM, Parzych J et al. Stream transport and retention of environmental DNA pulse releases in relation to hydrogeomorphic scaling factors. Environmental Science & Technology, 2019, 53(12): 6640-6649. DOI:10.1021/acs.est.8b06829 |
| [83] |
Pont D, Rocle M, Valentini A et al. Environmental DNA reveals quantitative patterns of fish biodiversity in large rivers despite its downstream transportation. Scientific Reports, 2018, 8: 10361. DOI:10.1038/s41598-018-28424-8 |
| [84] |
Carraro L, Stauffer JB, Altermatt F. How to design optimal eDNA sampling strategies for biomonitoring in river networks. Environmental DNA, 2021, 3(1): 157-172. DOI:10.1002/edn3.137 |
| [85] |
Thalinger B, Kirschner D, Pütz Y et al. Lateral and longitudinal fish environmental DNA distribution in dynamic riverine habitats. Environmental DNA, 2021, 3(1): 305-318. DOI:10.1002/edn3.171 |
| [86] |
Laporte M, Bougas B, Côté G et al. Caged fish experiment and hydrodynamic bidimensional modeling highlight the importance to consider 2D dispersion in fluvial environmental DNA studies. Environmental DNA, 2020, 2(3): 362-372. DOI:10.1002/edn3.88 |
| [87] |
Yang HL, Zhang H, Wu JM et al. Simulating the impacts of parallel samples on the estimations of upstream-to-downstream watershed biological information flow. Acta Ecologica Sinica, 2022, 42(13). [杨海乐, 张辉, 吴金明等. 平行样对自上游到下游的流域生物信息流估算的影响: 来自模拟计算的结果. 生态学报, 2022, 42(13). DOI:10.5846/stxb202104271109] |
| [88] |
Zhang S, Lu Q, Wang YY et al. Assessment of fish communities using environmental DNA: Effect of spatial sampling design in lentic systems of different sizes. Molecular Ecology Resources, 2020, 20(1): 242-255. DOI:10.1111/1755-0998.13105 |
| [89] |
Jeunen GJ, Lamare MD, Knapp M et al. Water stratification in the marine biome restricts vertical environmental DNA (eDNA) signal dispersal. Environmental DNA, 2020, 2(1): 99-111. DOI:10.1002/edn3.49 |
| [90] |
Koziol A, Stat M, Simpson T et al. Environmental DNA metabarcoding studies are critically affected by substrate selection. Molecular Ecology Resources, 2019, 19(2): 366-376. DOI:10.1111/1755-0998.12971 |
| [91] |
van der Heyde M, Bunce M, Wardell-Johnson G et al. Testing multiple substrates for terrestrial biodiversity monitoring using environmental DNA metabarcoding. Molecular Ecology Resources, 2020, 20(3): 10. DOI:10.1111/1755-0998.13148 |
| [92] |
Sakata MK, Yamamoto S, Gotoh RO et al. Sedimentary eDNA provides different information on timescale and fish species composition compared with aqueous eDNA. Environmental DNA, 2020, 2(4): 505-518. DOI:10.1002/edn3.75 |
| [93] |
Valentin RE, Kyle KE, Allen MC et al. The state, transport, and fate of aboveground terrestrial arthropod eDNA. Environmental DNA, 2021, 3(6): 1081-1092. DOI:10.1002/edn3.229 |
| [94] |
Eichmiller JJ, Miller LM, Sorensen PW. Optimizing techniques to capture and extract environmental DNA for detection and quantification of fish. Molecular Ecology Resources, 2016, 16(1): 56-68. DOI:10.1111/1755-0998.12421 |
| [95] |
Li J, Lawson Handley LJ, Read DS et al. The effect of filtration method on the efficiency of environmental DNA capture and quantification via metabarcoding. Molecular Ecology Resources, 2018. DOI:10.1111/1755-0998.12899 |
| [96] |
Majaneva M, Diserud OH, Eagle SHC et al. Environmental DNA filtration techniques affect recovered biodiversity. Scientific Reports, 2018, 8: 4682. DOI:10.1038/s41598-018-23052-8 |
| [97] |
Djurhuus A, Pitz K, Sawaya NA et al. Evaluation of marine zooplankton community structure through environmental DNA metabarcoding. Limnology and Oceanography, Methods, 2018, 16(4): 209-221. DOI:10.1002/lom3.10237 |
| [98] |
He XP, Sutherland TF, Abbott CL. Improved efficiency in eDNA metabarcoding of benthic metazoans by sieving sediments prior to DNA extraction. Environmental DNA, 2021, 3(4): 716-726. DOI:10.1002/edn3.172 |
| [99] |
Goldberg CS, Turner CR, Deiner K et al. Critical considerations for the application of environmental DNA methods to detect aquatic species. Methods in Ecology and Evolution, 2016, 7(11): 1299-1307. DOI:10.1111/2041-210x.12595 |
| [100] |
Yamanaka H, Minamoto T, Matsuura J et al. A simple method for preserving environmental DNA in water samples at ambient temperature by addition of cationic surfactant. Limnology, 2017, 18(2): 233-241. DOI:10.1007/s10201-016-0508-5 |
| [101] |
Sales NG, Wangensteen OS, Carvalho DC et al. Influence of preservation methods, sample medium and sampling time on eDNA recovery in a neotropical river. Environmental DNA, 2019, 1(2): edn3.14. DOI:10.1002/edn3.14 |
| [102] |
Casey JM, Ransome E, Collins AG et al. DNA metabarcoding marker choice skews perception of marine eukaryotic biodiversity. Environmental DNA, 2021, 3(6): 1229-1246. DOI:10.1002/edn3.245 |
| [103] |
Giebner H, Langen K, Bourlat SJ et al. Comparing diversity levels in environmental samples: DNA sequence capture and metabarcoding approaches using 18S and COI genes. Molecular Ecology Resources, 2020, 20(5): 1333-1345. DOI:10.1111/1755-0998.13201 |
| [104] |
Zhang S, Zhao JD, Yao M. A comprehensive and comparative evaluation of primers for metabarcoding eDNA from fish. Methods in Ecology and Evolution, 2020, 11(12): 1609-1625. DOI:10.1111/2041-210x.13485 |
| [105] |
Sommeria-Klein G, Zinger L, Coissac E et al. Latent Dirichlet Allocation reveals spatial and taxonomic structure in a DNA-based census of soil biodiversity from a tropical forest. Molecular Ecology Resources, 2020, 20(2): 371-386. DOI:10.1111/1755-0998.13109 |
| [106] |
Elbrecht V, Braukmann TWA, Ivanova NV et al. Validation of COI metabarcoding primers for terrestrial arthropods. PeerJ, 2019, 7: e7745. DOI:10.7717/peerj.7745 |
| [107] |
Collins RA, Bakker J, Wangensteen OS et al. Non-specific amplification compromises environmental DNA metabarcoding with COI. Methods in Ecology and Evolution, 2019, 10(11): 1985-2001. DOI:10.1111/2041-210x.13276 |
| [108] |
Luo JH, Tao Y, Xing P et al. Mini-review: Advances of metagenomics research for lake microbiomes. J Lake Sci, 2020, 32(1): 271-280. [罗建桦, 陶晔, 邢鹏等. 湖泊微生物宏基因组学研究进展. 湖泊科学, 2020, 32(1): 271-280. DOI:10.18307/2020.0125] |
| [109] |
Dopheide A, Xie D, Buckley TR et al. Impacts of DNA extraction and PCR on DNA metabarcoding estimates of soil biodiversity. Methods in Ecology and Evolution, 2019, 10(1): 120-133. DOI:10.1111/2041-210x.13086 |
| [110] |
Armbrecht L, Herrando-Pérez S, Eisenhofer R et al. An optimized method for the extraction of ancient eukaryote DNA from marine sediments. Molecular Ecology Resources, 2020, 20(4): 906-919. DOI:10.1111/1755-0998.13162 |
| [111] |
Hermans SM, Buckley HL, Lear G. Optimal extraction methods for the simultaneous analysis of DNA from diverse organisms and sample types. Molecular Ecology Resources, 2018, 18(3): 557-569. DOI:10.1111/1755-0998.12762 |
| [112] |
Ravindran S. Turning discarded DNA into ecology gold. Nature, 2019, 570(7762): 543-545. DOI:10.1038/d41586-019-01987-w |
| [113] |
Nichols RV, Vollmers C, Newsom LA et al. Minimizing polymerase biases in metabarcoding. Molecular Ecology Resources, 2018, 18. DOI:10.1111/1755-0998.12895 |
| [114] |
Antich A, Palacin C, Wangensteen OS et al. To denoise or to cluster, that is not the question: Optimizing pipelines for COI metabarcoding and metaphylogeography. BMC Bioinformatics, 2021, 22(1): 177. DOI:10.1186/s12859-021-04115-6 |
| [115] |
Kopylova E, Navas-Molina JA, Mercier C et al. Open-source sequence clustering methods improve the state of the art. mSystems, 2016, 1(1): e00003-e00015. DOI:10.1128/mSystems.00003-15 |
| [116] |
Peng XY, Dorman KS. AmpliCI: A high-resolution model-based approach for denoising Illumina amplicon data. Bioinformatics, 2020, 36(21): 5151-5158. DOI:10.1093/bioinformatics/btaa648 |
| [117] |
Yan HG, Dong ZL, Ma TT et al. Detection and biomass assessment of Procypris rabaudi based on environmental DNA. Journal of Fisheries of China, 2022, 46(6): 1018-1026. [闫卉果, 董智玲, 马婷婷等. 基于环境DNA的岩原鲤检测及生物量评估. 水产学报, 2022, 46(6): 1018-1026.] |
| [118] |
Capo E, Spong G, Koizumi S et al. Droplet digital PCR applied to environmental DNA, a promising method to estimate fish population abundance from humic-rich aquatic ecosystems. Environmental DNA, 2021, 3(2): 343-352. DOI:10.1002/edn3.115 |
| [119] |
Shen YJ, Hubert N, Huang Y et al. DNA barcoding the ichthyofauna of the Yangtze River: Insights from the molecular inventory of a mega-diverse temperate fauna. Molecular Ecology Resources, 2019, 19(5): 1278-1291. DOI:10.1111/1755-0998.12961 |
| [120] |
Wang M, Yuan Y, Yu HY et al. Construction of barcode library of freshwater Macroinvertebrate in China. Environmental Monitoring of China, 2022, 38(1): 36-44. [王萌, 苑艺, 于海燕等. 中国淡水大型底栖无脊椎动物条形码数据库构建. 中国环境监测, 2022, 38(1): 36-44.] |
| [121] |
Plante F, Bourgault P, Dubois Y et al. Environmental DNA as a detection and quantitative tool for stream-dwelling salamanders: A comparison with the traditional active search method. Environmental DNA, 2021, 3(6): 1128-1141. DOI:10.1002/edn3.233 |
| [122] |
Allen MC, Nielsen AL, Peterson DL et al. Terrestrial eDNA survey outperforms conventional approach for detecting an invasive pest insect within an agricultural ecosystem. Environmental DNA, 2021, 3(6): 1102-1112. DOI:10.1002/edn3.231 |
| [123] |
Berry O, Jarman S, Bissett A et al. Making environmental DNA (eDNA) biodiversity records globally accessible. Environmental DNA, 2021, 3(4): 699-705. DOI:10.1002/edn3.173 |
| [124] |
Sehnal L, Brammer-Robbins E, Wormington AM et al. Microbiome composition and function in aquatic vertebrates: Small organisms making big impacts on aquatic animal health. Frontiers in Microbiology, 2021, 12: 567408. DOI:10.3389/fmicb.2021.567408 |
| [125] |
Sun FL, Wang CZ, Chen LJ et al. The intestinal bacterial community of healthy and diseased animals and its association with the aquaculture environment. Applied Microbiology and Biotechnology, 2020, 104(2): 775-783. DOI:10.1007/s00253-019-10236-z |
| [126] |
Tong Q, Cui LY, Du XP et al. Comparison of gut microbiota diversity and predicted functions between healthy and diseased captive Rana dybowskii. Frontiers in Microbiology, 2020, 11: 2096. DOI:10.3389/fmicb.2020.02096 |
| [127] |
Sun FL, Xu ZT. Significant differences in intestinal microbial communities in aquatic animals from an aquaculture area. Journal of Marine Science and Engineering, 2021, 9(2): 104. |
| [128] |
Kuang TX, He AY, Lin YF et al. Comparative analysis of microbial communities associated with the gill, gut, and habitat of two filter-feeding fish. Aquaculture Reports, 2020, 18: 100501. DOI:10.1016/j.aqrep.2020.100501 |
| [129] |
Zhou L, Qu YY, Qin J et al. Deep insight into bacterial community characterization and relationship in the pond water, sediment and the gut of shrimp (Penaeus japonicus). Aquaculture, 2021, 539: 736658. DOI:10.1016/j.aquaculture.2021.736658 |
| [130] |
Zhang XC, Li XH, Lu JQ et al. Quantifying the importance of external and internal sources to the gut microbiota in juvenile and adult shrimp. Aquaculture, 2021, 531: 735910. DOI:10.1016/j.aquaculture.2020.735910 |
| [131] |
Trujillo-González A, Villacorta-Rath C, White NE et al. Considerations for future environmental DNA accreditation and proficiency testing schemes. Environmental DNA, 2021, 3(6): 1049-1058. DOI:10.1002/edn3.243 |
| [132] |
Maasri A, Jaehnig SC, Adamescu MC et al. A global agenda for advancing freshwater biodiversity research. Ecology Letters, 2022, 25(2): 255-263. DOI:10.1111/ele.13931 |
| [133] |
Michener WK, Jones MB. Ecoinformatics: Supporting ecology as a data-intensive science. Trends in Ecology & Evolution, 2012, 27(2): 85-93. DOI:10.1016/j.tree.2011.11.016 |
 2023, Vol. 35
2023, Vol. 35 

