(2: 中国科学院青藏高原研究所高寒生态重点实验室, 北京 100101)
(2: Key Laboratory of Alpine Ecology, Institute of Tibetan Plateau Research, Chinese Academy of Sciences, Beijing 100101, P. R. China)
湖泊沉积具有分布广泛、信息丰富、记录连续的特点,可为气候环境及生态系统演化提供良好的历史档案,因而是研究过去全球变化的重要途径之一[1-2]. 通过湖泊沉积记录研究气候环境演化及生态系统响应的工作已广泛开展,研究者通过对不同类型代用指标的分析以重建过去全球变化[3],已取得较多研究成果[1]. 随着湖泊沉积与环境演化研究工作的不断展开,传统方法逐渐难以满足学科深入发展的需求,新的技术不断被引入研究中[3-4],针对湖泊沉积物的DNA分析便为相关研究带来了机遇与动力. 与此同时,作为水生生态系统的组成部分,湖泊具有重要的生态服务功能,而近年来气候变化和人类活动对湖泊生态系统产生了显著影响[5-6],湖泊生态系统普遍面临来自外界的压力[7]. 多重压力作用下,湖泊综合状态可能从根本上发生改变[8-9],而不同区域湖泊状态响应人类活动与气候环境变化的过程、速度具有差异[10],由此导致的生态效应也并不相同,部分湖泊生态系统服务功能急剧退化. 因此利用湖泊沉积物系统研究不同时间尺度上湖泊响应人类活动与气候环境变化的过程、机制,以应对当今湖泊生态系统的可能转变愈加重要,古湖沼学研究亦需要更丰富的信息与多维的视角以弥补传统方法的不足. 分子生物学的迅速发展无疑为上述研究提供了新的机遇,随着学科交叉的发展与创新,高通量测序、荧光定量PCR等分子生物技术逐渐被应用于湖泊沉积物DNA中,为气候环境变化及生态系统响应等研究作出重要贡献.
古DNA分析是一个年轻但发展迅速的技术,自1980s研究者从已灭绝生物遗骸中提取DNA进行开创性研究以来[11],古DNA研究逐渐成为十分活跃的领域,在人类迁移、遗传关系、文明演化、动植物的驯化历史及古生物群落结构组成等方面取得了一系列进展[12],有效填补了现代DNA研究时间跨度较短的不足. 作为记录生物遗传信息的重要载体,DNA在古湖沼学研究中的潜力也逐渐被开发出来,Coolen等最早于1998年在加拿大Mahoney湖沉积物中针对硫氧化细菌DNA片段开展了研究[13],自此针对遗传物质的分析逐渐被应用到更多湖泊沉积记录中,为古气候、古环境及古生态研究提供了丰富的生物信息,取得了较新的科学认识.
湖泊沉积物中的DNA无疑为古湖沼学研究提供了更开阔的视野. 由于部分生物不能在湖泊沉积物中保留可被鉴定的形态学特征,DNA便成为生物重要的记录与档案之一[14]. 此外,沉积物中的古DNA可记录长尺度生态系统演化过程,与其他指标结合分析,可揭示气候变化与人类活动等多重因素驱动下的湖泊生态系统演化机制,并借助对气候环境变化敏感的生物群落开发新的气候环境代用指标. 本文以保存在湖泊沉积物中的DNA为对象,介绍其保存特点与影响因素,并以湖泊沉积物DNA在气候环境变化及生态系统响应研究中的应用进展为重点进行讨论,就其目前面临的挑战做出总结,简单展望未来的发展方向.
1 湖泊沉积物中的DNA湖泊沉积物蕴藏着生活或曾经生活在水体及流域的生物遗传信息[15],DNA保存程度与生物种类有密切关系. 在湖泊沉积物中,不同生物的DNA具有不同保存形式,既可保存在细胞内部,也可存在于细胞外部,与有机质或矿物基质结合[16],因此不同保存形式会对DNA产生影响. 例如,硅藻、沟鞭藻等生物具有较为坚硬的细胞壁[17],部分蓝藻在休眠期可产生厚壁孢子,一些原生动物可产生繁殖体[18],这些特性使胞内遗传物质得到物理保护. 尽管保存在细胞内部的生物DNA会在提取过程中遇到困难,但一般认为已知可产生刚性细胞壁的类群也可在沉积物DNA分析中被发现[16]. 某些细胞外部的生物DNA可以与沉积物中的矿物和有机大分子结合,从而抑制核酸酶降解[19-20]. 相比之下,一些水生生物DNA因缺乏物理保护,如隐藻DNA仅被保存在细胞膜中,可能通过组织脱落、废物代谢排出生物体[16],易受外界影响,在湖泊沉积物中保存状况较差[17]. 不同生物的遗传物质保存具有差异[15],而这种差异可能会对后续分析产生影响,值得慎重考虑.
除了生物本身的特性,湖泊水文特征与沉积物理化特征、沉积物年龄及沉积过程也会影响DNA的保存. 水文条件封闭的湖泊具有保存沉积物DNA的良好条件,其受到外部扰动较小,沉积物形成后的垂直运动与湖水动力作用较弱,DNA保存环境较稳定[16]. 深水湖泊比浅水湖泊更易产生稳定层节,悬浮及再沉积过程不易发生,而缺氧环境亦有利于生物DNA的保存[19]. 湖泊沉积物的组成、理化性质及孔隙水特征直接影响DNA,尤其是胞外DNA的保存[21]. 低温、缺氧、干燥、高盐度的环境有利于DNA的保存[22],其中温度、含氧程度不仅直接影响DNA保存,更间接影响核酸酶及微生物对DNA的分解与扰动过程[23],前人研究表明沉积物中微生物在降解有机质时会消耗氧气以创造出缺氧或无氧条件,弱化生物扰动,有利于遗传物质的保存[24]. 寒冷、干旱地区的湖泊沉积物DNA具有相对良好的保存条件,但热带及温带地区湖泊沉积物中成功进行的古DNA分析表明,只要条件适宜,温暖、湿润地区的湖泊沉积物亦可利用DNA分析进行相关研究[25-26].
年代较老的湖泊沉积物所保留的DNA含量较低,易被降解成较小片段[23],而DNA损伤与湖泊沉积物年龄也有关,沉积物老化往往导致DNA损伤增加(双链断裂、碱基错配、DNA交联损伤),造成后续分析的困难. 但年代限制并不绝对,Golenberg等从距今17百万~20百万年的古老湖相沉积物中提取DNA并对光合作用基因序列进行了扩增与测序[27],表明在良好埋藏条件下湖泊沉积物中古DNA的长时间保存是有可能的. 古生物死亡后,遗体成为沉积物的一部分,随着沉积物积累而被压实,从而允许研究者利用地质年代方法建立古生物记录[28]. 沉积物形成的过程有可能对生物DNA造成影响,水解、酶解、氧化及外部扰动等过程将直接造成DNA双螺旋主链断裂及降解[23],沉积物形成后可能的悬浮、再沉积过程也有可能扰动古DNA,并将现代DNA带入湖泊沉积物. 此外,遗传物质在沉积物中的迁移往往使胞外DNA分析结果变得复杂,在一些洞穴沉积物中可观察到DNA在垂直方向的迁移或浸出[29]. 但前人研究表明湖泊沉积物中DNA迁移和浸出可能性较小[30],许多湖泊沉积记录中进行的古DNA研究都是有力的证据[31-32]. 这正是湖泊沉积物保存DNA的优势所在:湖泊沉积物中水的重力渗透很难发生,当沉积物被压实时,孔隙水垂直平流十分微弱,沉积物中的物质得以被固定在基质中,胞外DNA附着在固相沉积物(如颗粒、微粒有机物)上从而被保护[30].
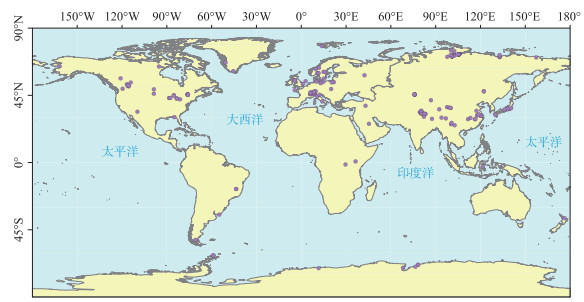
由湖泊沉积物中DNA上述特点可知,选择适宜的研究对象进行古DNA研究至关重要,研究前需要综合考虑区域环境、沉积物特点及目标生物DNA特征[17]. 我国湖泊数量众多、类型多样且分布广泛,许多湖泊具有保存沉积物DNA的良好条件,因而吸引了研究者的广泛关注与兴趣. 在世界范围内,针对湖泊沉积物中生物DNA的研究也已广泛开展[16](图 1),主要研究如表 1所示,获得了许多较有价值的研究成果,并表现出较大的发展潜力.
|
图 1 近年已开展沉积物DNA研究的湖泊 Fig.1 The distribution of lakes that have conducted sediment DNA research |
| 表 1 针对湖泊沉积物DNA的部分研究 Tab. 1 Research on DNA in lake sediments over the past few years |
湖泊沉积物中保存着大量的生物化石,通过孢粉、摇蚊、硅藻、介形虫等代用指标分析可开展古气候、古环境及古生态重建工作[65]. 传统生物指标主要建立在形态学鉴定的基础上,然而形态学方法鉴定分辨率较低,通过DNA记录可有效提高鉴定分辨率. 前人研究表明,湖泊沉积物古DNA分析结果与形态学鉴定结果具有良好一致性,DNA分析可将生物鉴定水平提升至种、亚种[16],来自遗传学水平的信息可揭示形态学鉴定所不能观察到的种间和种内变异[31]. 更重要的是,许多不能以化石形式保存在湖泊沉积物中的生物类群(例如微生物和多数浮游植物)往往也蕴含了丰富的气候环境信息,它们难以通过传统方法获得,却可通过DNA记录被鉴定[17],对没有脂类化合物和化石记录的病毒来说,古DNA分析更独具优势[66].
传统古生态研究集中关注特定生物种群,而单一种群指标难以解释群落变化所造成的生物多样性和生物网络变化,更难以揭示生态系统演化的关键驱动要素与机制[67]. 此外,群落结构不仅影响生态系统抵抗力稳定性与恢复力稳定性,还可影响生态系统弹性[64]. 因此,湖泊沉积物中的DNA记录填补了长尺度古生态数据缺乏的不足,在提供遗传信息的同时关注不同生物类群,以群落视角记录生态系统演化[68].
DNA分析不仅拓展了生物类群的研究范围,还可对重要的功能基因进行研究,如通过叶绿体rbcL基因序列及氨氧化古菌amoA基因序列[48, 69],可获得更多关于湖泊及流域物质循环的信息;而微囊藻毒素编码基因有助于揭示藻类群落中有毒蓝藻的动态及演变机制[70]. 这是传统古气候、古环境研究较少关注的内容,而DNA记录可为此提供来自遗传学视角的信息[66]. 例如Coolen等利用湖泊沉积物中的脂类物质追踪了全新世以来藻类群落演变,古DNA分析表明脂类物质来自6种不同基因型的藻类[71],而不同藻类基因丰度变化受控于湖泊环境的变化,这是单独使用生物标志化合物指标难以获得的信息.
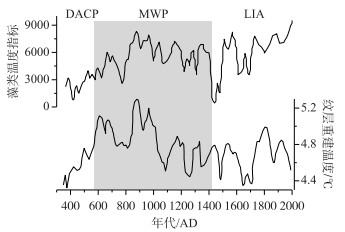
3 湖泊沉积物DNA分析的应用 3.1 重建古气候与古环境湖泊中部分生物对气候环境变化十分敏感(如某些浮游植物与微生物),因此具有记录气候环境变化的能力,是重建古气候、古环境的重要载体[16]. 古湖沼学研究中常通过样本训练集和转换函数的建立以推断过去的气候及环境条件,而湖泊沉积物中的DNA允许研究者关注更广泛的生物类群,尤其是无法通过化石被记录的生物,为定量、半定量重建古气候及古环境带来新的机遇. Li等通过青海湖沉积物中浮游植物古DNA记录重建了过去18500 a青海湖气候及环境演变历史,基于浮游植物的不同温度适应范围,将其划分为喜寒和喜暖两种类别,以其比值定义了藻类温度指标PI(phytoplankton index)[49],通过该指标重建的温度变化与An等重建的夏季风记录吻合良好[72]. 相比其他温度记录,浮游植物对温度变化更为敏感,因此PI指标能记录小幅度的温度波动. 为了进一步验证浮游植物温度指标,研究者将其应用于库赛湖沉积物的古DNA记录中,结果表明库赛湖PI指标所记录的温度变化与通过沉积纹层重建的温度记录吻合较好(图 2)[73].
|
图 2 库赛湖PI指标与纹层温度指标的对比(修改自文献[49-50]) Fig.2 The comparison of temperature record from phytoplankton index and varve-based paleo-temperature record in Lake Kusai (modified from literatures [49-50]) |
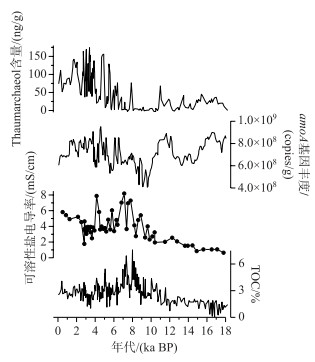
湖泊沉积物中的古DNA不仅能够追踪特定的生物类群,更可从功能基因的视角记录气候环境变化. 氨氧化古菌在湖泊及流域氮循环过程中扮演着重要角色,前人研究表明氨氧化古菌amoA基因丰度受盐度、pH、氨浓度、温度、营养状况、水深等湖沼条件的影响[74-81],而其与环境因素间的相关性为重建古环境提供了可能. Yang等基于氨氧化古菌amoA基因丰度与环境因素的关联重建了青海湖1.8万年来的营养水平与盐度的变化历史,其研究表明较强的东亚夏季风和充沛降水导致的湖泊高营养水平会抑制氨氧化古菌生长,而干燥和寒冷导致的低营养水平则促进其生长[48]. 因此,氨氧化古菌DNA记录具有重建湖泊营养水平、盐度及水位变化的能力,古菌DNA重建的湖泊营养水平与沉积物地球化学记录有较好一致性(图 3).
|
图 3 青海湖氨氧化古菌与环境指标的对比(修改自文献[48]) Fig.3 The comparison of the abundance of amoA-encoding archaea (AEA) from DNA and other proxies in Lake Qinghai (modified from literature [48]) |
古DNA能够以比形态学鉴定更高的分辨率广泛追踪不同生物类群,为研究陆地、水生生态系统对气候环境变化及人类活动的响应提供了更开阔的视野,有助于揭示生态系统演变的关键驱动要素与机制[16].
3.2.1 生态系统对气候环境变化的响应在缺乏长期监测数据的情况下,通过沉积记录研究湖泊及流域生态系统响应过去气候环境变化过程具有重要的科学与现实意义. Hou等利用DGGE结合克隆测序技术研究了青藏高原库赛湖浮游植物群落演变[50],沉积物DNA记录与古气候、古环境指标的对比分析表明浮游植物可敏感响应气候及湖泊状态变化,聚球藻和球等鞭金藻两种主要类群相对丰度均受亚洲季风影响:聚球藻对营养水平变化反应快,适合生存于高盐分、高营养水平的环境中,球等鞭金藻则与之相反,而季风通过影响外源输入改变湖泊营养水平与盐度,进而驱动库赛湖浮游植物群落的演变. 笔者通过沉积物DNA记录研究了位于季风边缘区高山湖泊公海中的浮游植物群落,结果亦表明浮游植物群落演变受到东亚夏季风影响,主要浮游植物聚球藻(Synechococcus sp.)与受控于东亚夏季风的流域化学风蚀指数(CIA)具有良好一致性[82].
微生物群落也可以敏感响应气候及环境变化,因而成为记录生态系统响应的良好载体. Ahmed等利用宏基因组测序与古菌细胞膜脂质对瑞典南部湖泊沉积物中的古菌群落进行了研究,结果表明古菌群落结构和多样性与水温、湖冰覆盖时间及湖泊沉积过程等因素有关,新仙女木至全新世过渡时期古菌多样性的显著增加与湖泊沼泽化有关[83]. 结合有机质含量及水温、脂类生物标记物等记录,研究者分析了古菌群落对气候和湖泊状态变化的不同响应过程及机制,发现古菌群落结构的微小变化虽然与气候有关,但受湖泊沼泽化的影响更明显. 湖泊中细菌群落在有机质分解和碳、氮、磷等主要元素的生物地球化学循环中起着关键作用,Zhang等使用高通量测序技术和冗余分析揭示了博斯腾湖沉积物中细菌群落的时空变化及其与环境因素间的关联,研究了细菌群落在湖泊营养程度和盐度、温度共同影响下的演变及机制[44]. 其研究表明博斯腾湖中细菌群落存在明显的季节变化且具有空间特异性,温度和营养状况是影响细菌群落结构的主要因素,细菌可通过调节群落结构以应对外界环境变化.
近年来真核微生物群落逐渐受到研究者的关注[84],真核微生物在生物地球化学循环过程中发挥着重要作用[85],将其纳入古生态学研究有助于深入理解生态系统演化及机制. Kisand等以拉脱维亚东部的Lielais Svēti n, u湖为对象,通过古DNA记录研究了过去14500 a以来真核生物多样性变化,结合孢粉、烧失量、炭屑等记录,重建了湖泊及周围陆地生态系统演化历史. 研究表明中全新世和过去2000 a生物多样性显著高于其他时期[62]. 农作物花粉数目及炭屑含量的增加表明近2000 a来人类活动对生态系统产生了强烈影响,造成生物多样性增加. 相比之下,中全新世生物多样性的增加则与温度升高、气候变暖有关. 在群落组成方面,中全新世以真菌和高等植物为主,过去2000 a则以绿藻等浮游生物为主. 通过古DNA记录重建的真核微生物群落演变表明生物多样性增加可能由不同的原因导致,不同驱动因素对生物多样性变化具有差异化的影响,中全新世蓝藻数量的增加与当代气候变暖及人类活动影响下蓝藻水华的发生机制并不相同.
湖泊沉积物接受了来自流域的外源输入物质,因此也可记录陆地生态系统演化. Voldstad等研究了斯瓦尔巴特群岛北部Jodavannet湖沉积物中的古DNA,结合地球化学指标,分析了末次冰消期以来流域植被群落结构及生物多样性对气候和环境变化的响应,结果表明全新世以来区域植被群落趋于稳定且喜热植被明显增加[61]. 现代记录中喜热植物存在于流域之外的山谷中,表明喜热植物在全新世的分布范围比现代更广. 研究者认为在全球气候变暖的作用下,喜热植物的扩张可能会对北极地区竞争力较弱的喜寒物种构成威胁,造成生物多样性降低[61].
3.2.2 生态系统对人类活动的响应19世纪中叶以来,人类活动逐渐成为一种重要的地质营力,对自然环境及生态系统产生着深刻的影响[86]. 研究人类活动影响下生态系统与气候环境相互作用的过程,可为当前生态系统的保护与治理提供借鉴,以预测、应对多重压力作用下生态系统可能发生的突变. 然而历史生态数据的缺乏限制了研究者对上述问题的深入理解,湖泊沉积物中的DNA记录则为揭示气候、人类活动与生态系统间相互作用提供了新的方法.
DNA记录可从遗传学视角评估生态系统演化,深入研究生物遗传多样性演变机制. Lendvay等对比了湖泊沉积物中保存的云杉古DNA及其现代DNA,结合花粉化石进一步研究了全新世以来云杉遗传多样性演变,结果表明人类对流域森林植被的砍伐始于晚全新世并在二十世纪初达到最大规模,造成云杉遗传多样性显著降低[52]. 植树造林及砍伐森林对生态系统的影响是复杂的,Sjögren等通过苏格兰两个湖泊沉积物中的古DNA研究了流域植被群落演变,结果表明在误差范围内沉积物中外来树种DNA出现时间与区域大范围人为造林时间非常吻合[87]. 植物DNA记录揭示了1960-2010年间流域植被从灌木丛到针叶林的过渡,与历史记录中人类种植针叶植被的时间吻合. 此外,人为疏松表土在改造土壤的同时也加剧了流域侵蚀程度.
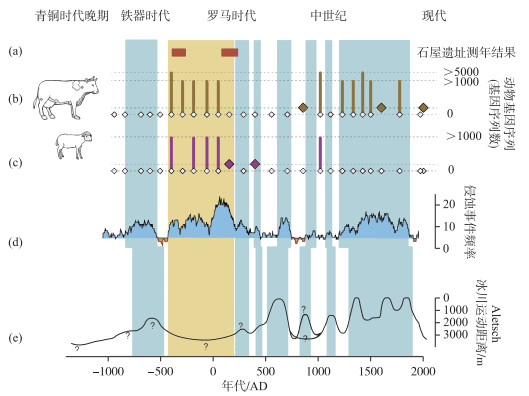
Giguet-Covex等以法国北部的Anterne湖为研究对象,基于湖泊沉积物中的生物DNA重建了新石器时代以来流域生态系统演化[37]. 结果表明流域侵蚀最严重的铁器时代晚期、罗马时期,植被DNA含量减少而牛羊等哺乳动物DNA含量增加(图 4),表明此时放牧活动十分活跃. 研究者认为人类活动是导致该时期区域侵蚀事件频发的主要因素:人类砍伐植被改变地表景观,放牧可压实土壤、分离土壤颗粒,植被覆盖率的降低与动物践踏加剧了流域侵蚀程度. 沉积物DNA记录证实了人类畜牧活动对流域生态系统的影响,揭示了不同气候环境条件下人类与生态系统间的复杂作用.
|
图 4 Anterne湖沉积物中动物DNA、侵蚀事件、冰川记录的对比(修改自文献[37]) Fig.4 The comparison of Bos sp. DNA and Ovis sp. DNA in sediments of Lake Anterne, erosion events and glacial records (modified from literature [37]) |
除了动植物DNA,湖泊沉积物中细菌DNA也可为研究人类活动与生态系统间的相互作用提供支持. Madeja研究了波兰东北部Łanskie湖泊沉积物中人类特异性拟杆菌HF 183的DNA序列,结合沉积物孢粉记录,探讨了人类活动对湖泊状态和流域植被的影响[40]. 研究结果表明这种人类特异性细菌DNA最早出现于公元650年左右,这一时期湖泊周围开始有人类定居,孢粉分析显示此时农作物花粉明显增加,而此前流域植被群落演变则受到人类活动以外其他因素的影响. 研究者认为保存在湖泊沉积物中的人类特异性遗传标记物可以提供有价值的信息以探索湖泊及流域生态系统对人类活动的响应(如植被演化、土壤侵蚀及养分变化等). 与植被孢粉记录相比,细菌DNA更能及时、准确记录人类活动历史及其对湖泊、流域生态系统的影响[40].
4 挑战与风险针对湖泊沉积物的DNA分析为研究气候环境及生态系统演化提供了新的方法,但现阶段仍存在若干技术问题. 我们认为当前存在的挑战主要来自以下几个方面:
1) 湖泊沉积物中DNA的保存与降解问题. 在一些年代较老的沉积物中,遗传物质已降解为较短片段,且DNA含量较低,部分生物的遗传信息甚至不能被保存,对开展后续分析造成了很大挑战. 即使DNA保存环境良好,生物扰动、湖泊水动力变化依然会损坏遗传物质. 因此,在进行古DNA研究之前,评估DNA降解情况以确保记录可靠性至关重要. 目前,对部分生物可通过其基因拷贝数与特异性标记物含量的相关性来评估DNA降解情况,长链烯酮含量与其产生者基因拷贝数之间的相关性常被用来评价古DNA的可靠性[12]. Hou等在青藏高原北部库赛湖的研究表明,沉积物中长链不饱和烯酮含量与其产生者的基因拷贝数具有良好相关性[50];Coolen等的研究表明晚全新世浮游植物古DNA含量与其产生的特异性长链不饱和烯酮有很好的相关性[71]. 此外,好氧氨氧化古菌及其产生的特征性标记物类异戊二烯、甲藻及其产生的甾醇间的相关性也可被用来评估古DNA的保存程度[15].
2) 湖泊沉积物DNA的提取问题. 目前湖泊沉积物中古DNA提取多采用土壤试剂盒提取样品总DNA,能否有效释放样品DNA并进行纯化是影响DNA提取的关键因素[12]. 不同湖泊沉积物样品的物理、化学性质存在差异,因此DNA提取方法与效率也并不相同,常规的DNA提取流程并不适用于不同类型的沉积样品及目标DNA. 如何高效、准确地提取不同性质样品中的目的DNA是值得重视的问题,因此需针对样品及目的DNA研究可行的实验方法.
3) 目的DNA的识别与潜在污染. 湖泊中沉积物不断与水体、生物进行着物质循环与交换,如何从动态的湖泊生态系统中识别出目标体系DNA及生物类群,从而避免后期实验污染及其他体系DNA的交叉干扰是至关重要的问题. 由于现代DNA在环境中普遍存在,应从采样开始就采取严格的预防措施以避免污染. 单一策略难以有效规避所有潜在污染源,如环境污染、交叉污染、基因提取和扩增时的可能污染等,多种方法的结合可以最大程度地避免污染以获得可靠信息[88-89]. 现有技术已提供一些良好的方法来确保实验的可靠性,如设置空白对照实验组、重复实验、DNA示踪等(表 2). 在数据处理阶段,当出现可疑的结果时,必须谨慎对待.
| 表 2 湖泊沉积物DNA分析的主要步骤、风险和可能的预防措施(修改自文献[16]) Tab. 2 Main analytical steps, risks and possible solutions for implementation of DNA analyses in lake sediments (modified from literature [16]) |
4) 学科交叉融合与创新的挑战. 目前针对生物DNA测序的研究方法与分析流程多是基于生物学研究需求所建立,而在学科交叉与融合的发展过程中,原有的生物学研究方法、分析流程与相关指标并不完全适合地理学的学科特征与研究需求. 随着未来学科交叉渗透的不断深入,在沉积物DNA研究中应当建立适宜的数据分析体系、指标以促进相关学科的发展.
5 展望与发展湖泊沉积物具有保存生物遗传信息的适宜条件,是进行DNA分析的良好对象. 湖泊沉积物中的DNA记录为气候环境变化与生态系统响应研究带来了新的机遇[16],扩大了研究生物的范围并提供丰富的信息,有助于研究微体生物化石不能记录的关键生物类群和重要生态过程,从遗传学层面揭示生态系统对气候环境变化及人类活动的响应过程. 我们认为未来针对湖泊沉积物中DNA的研究有望在以下几个方面取得发展:
1) 环境DNA分析结合复活生态学以评估外界环境变化对单个物种基因型和表型的影响. 大多数古DNA研究缺乏对基因型与表型的分析,尽管基因组学可提供相关研究机会,但种群水平分析的分辨率较低且难以研究表型和生物适应的具体变化[90]. 复活生态学与古DNA分析的结合则将种群遗传基因及表型转变与外在环境变化联系起来,研究气候环境变化和人类影响下生物遗传多样性变化的速率和幅度,阐明在面对多种压力时生物的适应过程.
2) 对生物入侵及机制的理解. 传统古湖沼学方法难以深入研究入侵物种对湖泊及流域生态系统的影响及机制. DNA分析可展现包括动物、植物和病原体等多种入侵生态系统的生物[91-92]. 此类研究不仅可以重建生物入侵历史,更可揭示其动力学机制以帮助研究者预测复杂情况下生物入侵引起的生态效应.
3) 对历史生物多样性及生物网络的研究. 针对生物多样性及生物网络的分析有助于深入研究生物群落间相互作用及其演变机制. 网络分析可识别生态系统中的重要生物,进而针对性地研究关键类群,并通过网络拓扑结构研究生物种内及种间关系[93]. 湖泊沉积物DNA所蕴含的生物信息使其具有洞察群落内部作用的潜力,而现有的古生态研究在多样性与生物网络方面较为匮乏.
4) 探索新的代用指标定量重建古气候、古环境演化历史. 古DNA记录允许研究者关注更广泛的生物群落以建立新的代用指标[94],如可以反映湖泊营养状态且DNA保存良好的纤毛虫[95-96]. 此外,古DNA记录揭示了部分隐藏在生物形态学背后的变异及遗传多样性[31],与传统形态学鉴定相结合,有助于提高转换函数准确度,更全面地重建气候、环境变化历史[16]. 目前该方向研究较少,或与古DNA的保存及定量分析等问题有关,但在未来有可能会帮助研究者获得更好的古气候、古环境代用指标.
5) 多组学整合分析以深入探索科学问题. 随着高通量测序技术的不断发展与完善,对不同层次、类型生物数据的获取、分析方法日趋成熟,而多组学相互促进、验证的研究方法也逐渐成为分子生物学的重要技术[97]. 通过研究生物体内不同组分的结构及特定条件下彼此间的相互作用,可分析外界干扰下生物内部应对过程与机制. 在利用湖泊沉积物开展生物地球化学研究的过程中,应当积极尝试代谢组、转录组、蛋白质组等多组学结合的研究方法,深入开展科学研究与探索.
湖泊沉积物中的DNA记录有助于解决许多令人兴奋的问题,将湖泊沉积物DNA分析与传统研究方法相结合,发展了气候环境变化及生态系统响应等研究的深度与广度,有利于学科交叉的发展与创新. 为了获得稳健可靠的结论,在研究过程中应当采取严格的实验操作,谨慎解析数据,并参考其他指标所提供的信息,根据科学问题及研究重点展开工作.
致谢: 感谢兰州大学博士研究生王强、吕飞亚、张姚以及硕士研究生刘言在本论文撰写过程中的有益讨论,特此致谢.
| [1] |
Shen J. Progress and prospect of palaeolimnology research in China. J Lake Sci, 2009, 21(3): 307-313. [沈吉. 湖泊沉积研究的历史进展与展望. 湖泊科学, 2009, 21(3): 307-313. DOI:10.18307/2009.0301] |
| [2] |
Wang SM, Zhang ZK. New progress of lake sediments and environmental changes research in China. Chinese Science Bulletin, 1999, 44(19): 1744-1754. [王苏民, 张振克. 中国湖泊沉积与环境演变研究的新进展. 科学通报, 1999, 44(19): 1744-1754. DOI:10.1007/BF02886151] |
| [3] |
Pu PM, Tu QY, Wang SM. Progress of limnology in China. Chinese Journal of Oceanology and Limnology, 1991, 9(3): 193-206. DOI:10.1007/BF02850745 |
| [4] |
Chen FH, Yu ZC, Yang ML et al. Holocene moisture evolution in arid central Asia and its out-of-phase relationship with Asian monsoon history. Quaternary Science Reviews, 2008, 27(3/4): 351-364. DOI:10.1016/j.quascirev.2007.10.017 |
| [5] |
Ho JC, Michalak AM, Pahlevan N. Widespread global increase in intense lake phytoplankton blooms since the 1980s. Nature, 2019, 574(7780): 667-670. DOI:10.1038/s41586-019-1648-7 |
| [6] |
Iestyn WR, Merchant CJ. Worldwide alteration of lake mixing regimes in response to climate change. Nature Geoscience, 2019, 12(4): 271-276. DOI:10.1038/s41561-019-0322-x |
| [7] |
Ormerod SJ, Dobson M, Hildrew AG et al. Multiple stressors in freshwater ecosystems. Freshwater Biology, 2010, 55: 1-4. DOI:10.1111/j.1365-2427.2009.02395.x |
| [8] |
Eastwood WJ, Roberts N, Lamb HF et al. Holocene environmental change in southwest Turkey: A palaeoecological record of lake and catchment-related changes. Quaternary Science Reviews, 1999, 18(4/5): 671-695. DOI:10.1016/S0277-3791(98)00104-8 |
| [9] |
Ye XC, Zhang Q, Liu J et al. Distinguishing the relative impacts of climate change and human activities on variation of streamflow in the Poyang Lake catchment, China. Journal of Hydrology, 2013, 494: 83-95. DOI:10.1016/j.jhydrol.2013.04.036 |
| [10] |
Ma RH, Duan HT, Hu CM et al. A half-century of changes in China's lakes: Global warming or human influence?. Geophysical Research Letters, 2010, 37(24). DOI:10.1029/2010gl045514 |
| [11] |
Higuchi RG, Wrischnik LA, Oakes E et al. Mitochondrial DNA of the extinct quagga: Relatedness and extent of postmortem change. Journal of Molecular Evolution, 1987, 25(4): 283-287. DOI:10.1007/BF02603111 |
| [12] |
Yan DN, Xu H, Hou WG. Application of modern molecular biotechnology in studying ancient DNA of lake sediments. Chinese Journal of Ecology, 2019, 38(7): 2200-2209. [严东娜, 徐海, 侯卫国. 现代分子生物技术在湖泊沉积物古DNA研究中的应用. 生态学杂志, 2019, 38(7): 2200-2209. DOI:10.13292/j.1000-4890.201907.009] |
| [13] |
Coolen MJ, Overmann J. Analysis of subfossil molecular remains of purple sulfur bacteria in a lake sediment. Applied and Environmental Microbiology, 1998, 64(11): 4513-4521. DOI:10.1128/AEM.64.11.4513-4521.1998 |
| [14] |
Schleper CA, Holben W, Klenk HP. Recovery of crenarchaeotal ribosomal DNA sequences from freshwater-lake sediments. Applied & Environmental Microbiology, 1997, 63(1): 321-323. DOI:10.1128/AEM.63.1.321-323.1997 |
| [15] |
Boere AC, Abbas B, Rijpstra WIC et al. Late-Holocene succession of dinoflagellates in an Antarctic fjord using a multiproxy approach: Paleoenvironmental genomics, lipid biomarkers and palynomorphs. Geobiology, 2009, 7(3): 265-281. DOI:10.1111/j.1472-4669.2009.00202.x |
| [16] |
Domaizon I, Winegardner A, Capo E et al. DNA-based methods in paleolimnology: New opportunities for investigating long-term dynamics of lacustrine biodiversity. Journal of Paleolimnology, 2017, 58(1): 1-21. DOI:10.1007/s10933-017-9958-y |
| [17] |
Capo E, Debroas D, Arnaud F et al. Is planktonic diversity well recorded in sedimentary DNA? Toward the reconstruction of past protistan diversity. Microbial Ecology, 2015, 70(4): 865-875. DOI:10.1007/s00248-015-0627-2 |
| [18] |
HHärnström K, Ellegaard M, Andersen TJ et al. Hundred years of genetic structure in a sediment revived diatom population. PNAS, 2011, 108(10): 4252-4257. DOI:10.1073/pnas.1013528108 |
| [19] |
Corinaldesi C, Beolchini F, Dell'anno A. Damage and degradation rates of extracellular DNA in marine sediments: Implications for the preservation of gene sequences. Molecular Ecology, 2008, 17(17): 3939-3951. DOI:10.1111/j.1365-294X.2008.03880.x |
| [20] |
Dell'Anno A, Stefano B, Danovaro R. Quantification, base composition, and fate of extracellular DNA in marine sediments. Limnology and Oceanography, 2002, 47(3): 899-905. DOI:10.4319/lo.2002.47.3.0899 |
| [21] |
Briggs AW, Stenzel U, Johnson PLF et al. Patterns of damage in genomic DNA sequences from a Neandertal. PNAS, 2007, 104(37): 14616-14621. DOI:10.1073/pnas.0704665104 |
| [22] |
Panieri G, Lugli S, Manzi V et al. Ribosomal RNA gene fragments from fossilized cyanobacteria identified in primary gypsum from the late Miocene, Italy. Geobiology, 2010, 8(2): 101-111. DOI:10.1111/j.1472-4669.2009.00230.x |
| [23] |
Hofreiter M, Serre D, Poinar HN et al. Ancient DNA. Nature Reviews Genetics, 2001, 2(5): 353-359. DOI:10.1038/35072071 |
| [24] |
Sobek S, Durisch-Kaiser E, Zurbrügg R et al. Organic carbon burial efficiency in lake sediments controlled by oxygen exposure time and sediment source. Limnology and Oceanography, 2009, 54(6): 2243-2254. DOI:10.4319/lo.2009.54.6.2243 |
| [25] |
Epp LS, Stoof KR, Trauth MH et al. Historical genetics on a sediment core from a Kenyan lake: Intraspecific genotype turnover in a tropical rotifer is related to past environmental changes. Journal of Paleolimnology, 2010, 43(4): 939-954. DOI:10.1007/s10933-009-9379-7 |
| [26] |
Yan DN, Xu H, Yang M et al. Responses of cyanobacteria to climate and human activities at Lake Chenghai over the past 100 years. Ecological Indicators, 2019, 104: 755-763. DOI:10.1016/j.ecolind.2019.03.019 |
| [27] |
Golenberg EM, Giannasi DE, Clegg MT et al. Chloroplast DNA sequence from a Miocene Magnolia species. Nature, 1990, 344(6267): 656-658. DOI:10.1038/344656a0 |
| [28] |
Parducci L, Bennett KD, Ficetola GF et al. Ancient plant DNA in lake sediments. New Phytologist, 2017, 214(3): 924-942. DOI:10.1111/nph.14470 |
| [29] |
Haile J, Holdaway R, Oliver K et al. Ancient DNA chronology within sediment deposits: Are paleobiological reconstructions possible and is DNA leaching a factor?. Molecular Biology and Evolution, 2007, 24(4): 982-989. DOI:10.1093/molbev/msm016 |
| [30] |
Anderson-Carpenter LL, McLachlan JS, Jackson ST et al. Ancient DNA from lake sediments: Bridging the gap between paleoecology and genetics. BMC Evolutionary Biology, 2011, 11: 30. DOI:10.1186/1471-2148-11-30 |
| [31] |
Stoof-Leichsenring KR, Epp LS, Trauth MH et al. Hidden diversity in diatoms of Kenyan Lake Naivasha: A genetic approach detects temporal variation. Molecular Ecology, 2012, 21(8): 1918-1930. DOI:10.1111/j.1365-294X.2011.05412.x |
| [32] |
Monchamp ME, Walser JC, Pomati F et al. Sedimentary DNA reveals cyanobacterial community diversity over 200 years in two perialpine lakes. Applied and Environmental Microbiology, 2016, 82(21): 6472-6482. DOI:10.1128/aem.02174-16 |
| [33] |
Limburg PA, Weider LJ. 'Ancient' DNA in the resting egg bank of a microcrustacean can serve as a palaeolimnological database. Proceedings of the Royal Society of London Series B: Biological Sciences, 2002, 269(1488): 281-287. DOI:10.1098/rspb.2001.1868 |
| [34] |
Reid VA, Carvalho GR, George DG et al. A technique for the molecular genetic analysis of Daphnia resting eggs from sub-recent lake sediments. Journal of Paleolimnology, 2002, 27(4): 481-486. DOI:10.1023/A:1020380918518 |
| [35] |
Matisoo-Smith E, Roberts K, Welikala N et al. Recovery of DNA and pollen from New Zealand lake sediments. Quaternary International, 2008, 184(1): 139-149. DOI:10.1016/j.quaint.2007.09.013 |
| [36] |
Xu ZH, Jiang XD, Wang GZ et al. DNA extraction, amplification and analysis of the 28S rRNA portion in sediment-buried copepod DNA in the Great Wall Bay and Xihu Lake, Antarctica. Journal of Plankton Research, 2011, 33(6): 917-925. DOI:10.1093/plankt/fbq165 |
| [37] |
Giguet-Covex C, Pansu J, Arnaud F et al. Long livestock farming history and human landscape shaping revealed by lake sediment DNA. Nature Communications, 2014, 5: 3211. DOI:10.1038/ncomms4211 |
| [38] |
Ficetola GF, Poulenard J, Sabatier P et al. DNA from lake sediments reveals long-term ecosystem changes after a biological invasion. Science Advances, 2018, 4(5): eaar4292. DOI:10.1126/sciadv.aar4292 |
| [39] |
Bissett A, Gibson JAE, Jarman SN et al. Isolation, amplification, and identification of ancient copepod DNA from lake sediments. Limnology and Oceanography: Methods, 2005, 3(12): 533-542. DOI:10.4319/lom.2005.3.533 |
| [40] |
Madeja J. A new tool to trace past human presence from lake sediments: The human-specific molecular marker Bacteroides strain HF 183. Journal of Quaternary Science, 2015, 30(4): 349-354. DOI:10.1002/jqs.2783 |
| [41] |
Liu YQ, Conrad R, Yao TD et al. Change of methane production pathway with sediment depth in a lake on the Tibetan Plateau. Palaeogeography, Palaeoclimatology, Palaeoecology, 2017, 474: 279-286. DOI:10.1016/j.palaeo.2016.06.021 |
| [42] |
Liu WZ, Yao L, Jiang XL et al. Sediment denitrification in Yangtze lakes is mainly influenced by environmental conditions but not biological communities. Science of the Total Environment, 2018, 616/617: 978-987. DOI:10.1016/j.scitotenv.2017.10.221 |
| [43] |
Zhang H, Wan ZW, Ding MJ et al. Inherent bacterial community response to multiple heavy metals in sediment from river-lake systems in the Poyang Lake, China. Ecotoxicology and Environmental Safety, 2018, 165: 314-324. DOI:10.1016/j.ecoenv.2018.09.010 |
| [44] |
Zhang L, Shen TT, Cheng Y et al. Temporal and spatial variations in the bacterial community composition in Lake Bosten, a large, brackish lake in China. Scientific Reports, 2020, 10: 304. DOI:10.1038/s41598-019-57238-5 |
| [45] |
Poulain AJ, Aris-Brosou S, Blais JM et al. Microbial DNA records historical delivery of anthropogenic mercury. The ISME Journal, 2015, 9(12): 2541-2550. DOI:10.1038/ismej.2015.86 |
| [46] |
Jiang HC, Dong HL, Yu BS et al. Microbial response to salinity change in Lake Chaka, a hypersaline lake on Tibetan Plateau. Environmental Microbiology, 2007, 9(10): 2603-2621. DOI:10.1111/j.1462-2920.2007.01377.x |
| [47] |
Coolen MJL, Talbot HM, Abbas BA et al. Sources for sedimentary bacteriohopanepolyols as revealed by 16S rDNA stratigraphy. Environmental Microbiology, 2008, 10(7): 1783-1803. DOI:10.1111/j.1462-2920.2008.01601.x |
| [48] |
Yang J, Jiang HC, Dong HL et al. Sedimentary archaeal AmoA gene abundance reflects historic nutrient level and salinity fluctuations in Qinghai Lake, Tibetan Plateau. Scientific Reports, 2015, 5: 18071. DOI:10.1038/srep18071 |
| [49] |
Li GY, Dong HL, Hou WG et al. Temporal succession of ancient phytoplankton community in Qinghai Lake and implication for paleo-environmental change. Scientific Reports, 2016, 6: 19769. DOI:10.1038/srep19769 |
| [50] |
Hou WG, Dong HL, Li GY et al. Identification of photosynthetic plankton communities using sedimentary ancient DNA and their response to late-Holocene climate change on the Tibetan Plateau. Scientific Reports, 2014, 4: 6648. DOI:10.1038/srep06648 |
| [51] |
Parducci L, Jorgensen T, Tollefsrud MM et al. Glacial survival of boreal trees in northern Scandinavia. Science, 2012, 335(6072): 1083-1086. DOI:10.1126/science.1216043 |
| [52] |
Lendvay B, Bálint M, Pál I et al. Plant macrofossils from lake sediment as the material to assess ancient genetic diversity: Did deforestation influence Norway spruce (Picea abies) in the South Carpathians?. Quaternary International, 2018, 477: 106-116. DOI:10.1016/j.quaint.2018.02.023 |
| [53] |
Randlett Mō, Coolen MJL, Stockhecke M et al. Alkenone distribution in Lake Van sediment over the last 270 ka: Influence of temperature and haptophyte species composition. Quaternary Science Reviews, 2014, 104: 53-62. DOI:10.1016/j.quascirev.2014.07.009 |
| [54] |
Pansu J, Giguet-Covex C, Ficetola GF et al. Reconstructing long-term human impacts on plant communities: An ecological approach based on lake sediment DNA. Molecular Ecology, 2015, 24(7): 1485-1498. DOI:10.1111/mec.13136 |
| [55] |
Alsos IG, Lammers Y, Yoccoz NG et al. Plant DNA metabarcoding of lake sediments: How does it represent the contemporary vegetation. PLoS One, 2018, 13(4): e0195403. DOI:10.1371/journal.pone.0195403 |
| [56] |
Epp LS, Gussarova G, Boessenkool S et al. Lake sediment multi-taxon DNA from North Greenland records early post-glacial appearance of vascular plants and accurately tracks environmental changes. Quaternary Science Reviews, 2015, 117(2): 152-163. DOI:10.1016/j.quascirev.2015.03.027 |
| [57] |
Alsos IG, Coissac E, Edwards M et al. Plant DNA in sediments: To which degree do they represent the flora?. Genome, 2015, 58(5): 186. |
| [58] |
Paus A, Boessenkool S, Brochmann C et al. Lake Store Finnsjøen-a key for understanding Lateglacial/early Holocene vegetation and ice sheet dynamics in the central Scandes Mountains. Quaternary Science Reviews, 2015, 121: 36-51. DOI:10.1016/j.quascirev.2015.05.004 |
| [59] |
Magyari EK, Major Á, Bálint M et al. Population dynamics and genetic changes of Picea abies in the South Carpathians revealed by pollen and ancient DNA analyses. BMC Evolutionary Biology, 2011, 11(1): 1-16. DOI:10.1186/1471-2148-11-66 |
| [60] |
Kyle M, Haande S, Sønstebø J et al. Amplification of DNA in sediment cores to detect historic Planktothrix occurrence in three Norwegian lakes. Journal of Paleolimnology, 2015, 53(1): 61-72. DOI:10.1007/s10933-014-9807-1 |
| [61] |
Voldstad LH, Alsos IG, Farnsworth WR et al. A complete Holocene lake sediment ancient DNA record reveals long-standing high Arctic plant diversity hotspot in northern Svalbard. Quaternary Science Reviews, 2020, 234: 106207. DOI:10.1016/j.quascirev.2020.106207 |
| [62] |
Kisand V, Talas L, Kisand A et al. From microbial eukaryotes to metazoan vertebrates: Wide spectrum paleo-diversity in sedimentary ancient DNA over the last~14, 500 years. Geobiology, 2018, 16(6): 628-639. DOI:10.1111/gbi.12307 |
| [63] |
Boessenkool S, McGlynn G, Epp LS et al. Use of ancient sedimentary DNA as a novel conservation tool for high-altitude tropical biodiversity. Conservation Biology, 2014, 28(2): 446-455. DOI:10.1111/cobi.12195 |
| [64] |
Savichtcheva O, Debroas D, Perga ME et al. Effects of nutrients and warming on Planktothrix dynamics and diversity: A palaeolimnological view based on sedimentary DNA and RNA. Freshwater Biology, 2015, 60(1): 31-49. DOI:10.1111/fwb.12465 |
| [65] |
Liang J, Hou JZ, Li D et al. Review on investigating changes in lake ecosystems using sedimentary pigments. Quaternary Sciences, 2016, 36(3): 630-645. [梁洁, 侯居峙, 李栋等. 沉积色素定量重建湖泊系统变化研究进展. 第四纪研究, 2016, 36(3): 630-645.] |
| [66] |
Coolen MJL. 7000 years of Emiliania huxleyi viruses in the Black Sea. Science, 2011, 333(6041): 451-452. DOI:10.1126/science.1200072 |
| [67] |
Davidson TA, Reid MA, Sayer CD et al. Palaeolimnological records of shallow lake biodiversity change: Exploring the merits of single versus multi-proxy approaches. Journal of Paleolimnology, 2013, 49(3): 431-446. DOI:10.1007/s10933-013-9696-8 |
| [68] |
Capo E, Debroas D, Arnaud F et al. Tracking a century of changes in microbial eukaryotic diversity in lakes driven by nutrient enrichment and climate warming. Environmental Microbiology, 2017, 19(7): 2873-2892. DOI:10.1111/1462-2920.13815 |
| [69] |
Willerslev E, Hansen AJ, Binladen J et al. Diverse plant and animal genetic records from Holocene and Pleistocene sediments. Science, 2003, 300(5620): 791-795. DOI:10.1126/science.1084114 |
| [70] |
Song H, Coggins LX, Reichwaldt ES et al. The importance of lake sediments as a pathway for microcystin dynamics in shallow eutrophic lakes. Toxins: Basel, 2015, 7(3): 900-918. DOI:10.3390/toxins7030900 |
| [71] |
Coolen MJL, Muyzer G, Rijpstra WIC et al. Combined DNA and lipid analyses of sediments reveal changes in Holocene haptophyte and diatom populations in an Antarctic lake. Earth and Planetary Science Letters, 2004, 223(1/2): 225-239. DOI:10.1016/j.epsl.2004.04.014 |
| [72] |
An ZS, Colman SM, Zhou WJ et al. Interplay between the Westerlies and Asian monsoon recorded in Lake Qinghai sediments since 32 ka. Scientific Reports, 2012, 2: 619. DOI:10.1038/srep00619 |
| [73] |
Liu XQ, Yu ZT, Dong HL et al. A less or more dusty future in the Northern Qinghai-Tibetan Plateau?. Scientific Reports, 2014, 4(1): 1-6. DOI:10.1038/srep06672 |
| [74] |
Yang J, Jiang HC, Dong HL et al. AmoA-encoding archaea and thaumarchaeol in the lakes on the northeastern Qinghai-Tibetan Plateau, China. Frontiers in Microbiology, 2013, 4: 329. DOI:10.3389/fmicb.2013.00329 |
| [75] |
Auguet JC, Casamayor EO. Partitioning of Thaumarchaeota populations along environmental gradients in high mountain lakes. FEMS Microbiology Ecology, 2013, 84(1): 154-164. DOI:10.1111/1574-6941.12047 |
| [76] |
Hugoni M, Etien S, Bourges A et al. Dynamics of ammonia-oxidizing archaea and bacteria in contrasted freshwater ecosystems. Research in Microbiology, 2013, 164(4): 360-370. DOI:10.1016/j.resmic.2013.01.004 |
| [77] |
Auguet JC, Nomokonova N, Camarero L et al. Seasonal changes of freshwater ammonia-oxidizing archaeal assemblages and nitrogen species in oligotrophic alpine lakes. Applied and Environmental Microbiology, 2011, 77(6): 1937-1945. DOI:10.1128/AEM.01213-10 |
| [78] |
Buckles LK, Villanueva L, Weijers JWH et al. Linking isoprenoidal GDGT membrane lipid distributions with gene abundances of ammonia-oxidizing Thaumarchaeota and uncultured crenarchaeotal groups in the water column of a tropical lake (Lake Challa, East Africa). Environmental Microbiology, 2013, 15(9): 2445-2462. DOI:10.1111/1462-2920.12118 |
| [79] |
Wu YC, Xiang Y, Wang JJ et al. Heterogeneity of archaeal and bacterial ammonia-oxidizing communities in Lake Taihu, China. Environmental Microbiology Reports, 2010, 2(4): 569-576. DOI:10.1111/j.1758-2229.2010.00146.x |
| [80] |
Pouliot J, Galand PE, Lovejoy C et al. Vertical structure of archaeal communities and the distribution of ammonia monooxygenase A gene variants in two meromictic High Arctic lakes. Environmental Microbiology, 2009, 11(3): 687-699. DOI:10.1111/j.1462-2920.2008.01846.x |
| [81] |
Auguet JC, Triadó-Margarit X, Nomokonova N et al. Vertical segregation and phylogenetic characterization of ammonia-oxidizing Archaea in a deep oligotrophic lake. The ISME Journal, 2012, 6(9): 1786-1797. DOI:10.1038/ismej.2012.33 |
| [82] |
Liu JB, Chen JH, Kandasamy S et al. A 14.7 Ka record of earth surface processes from the arid-monsoon transitional zone of China. Earth Surface Processes and Landforms, 2018, 43(3): 723-734. DOI:10.1002/esp.4265 |
| [83] |
Ahmed E, Parducci L, Unneberg P et al. Archaeal community changes in Lateglacial lake sediments: Evidence from ancient DNA. Quaternary Science Reviews, 2018, 181: 19-29. DOI:10.1016/j.quascirev.2017.11.037 |
| [84] |
Pawlowski J, Kelly-Quinn M, Altermatt F et al. The future of biotic indices in the ecogenomic era: Integrating (e)DNA metabarcoding in biological assessment of aquatic ecosystems. Science of the Total Environment, 2018, 637/638: 1295-1310. DOI:10.1016/j.scitotenv.2018.05.002 |
| [85] |
Dinsdale EA, Edwards RA, Hall D et al. Erratum: Functional metagenomic profiling of nine biomes. Nature, 2008, 455(7214): 830. DOI:10.1038/nature07346 |
| [86] |
Lotstein EL. Global climate change impacts in the United States: A state of knowledge report from the US global climate change research program. Journal of Geography, 2013, 112(4): 178. DOI:10.1080/00221341.2013.770905 |
| [87] |
Sjögren P, Edwards ME, Gielly L et al. Lake sedimentary DNA accurately records 20th Century introductions of exotic conifers in Scotland. The New Phytologist, 2017, 213(2): 929-941. DOI:10.1111/nph.14199 |
| [88] |
Schouten S, Hopmans EC, Pancost RD et al. Widespread occurrence of structurally diverse tetraether membrane lipids: Evidence for the ubiquitous presence of low-temperature relatives of hyperthermophiles. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(26): 14421-14426. DOI:10.1073/pnas.97.26.14421 |
| [89] |
Champlot S, Berthelot C, Pruvost M et al. An efficient multistrategy DNA decontamination procedure of PCR reagents for hypersensitive PCR applications. PLoS One, 2010, 5(9): e744. DOI:10.1371/journal.pone.0013042 |
| [90] |
Giguet-Covex C, Ficetola GF, Walsh K et al. New insights on lake sediment DNA from the catchment: Importance of taphonomic and analytical issues on the record quality. Scientific Reports, 2019, 9(1): 14676. DOI:10.1038/s41598-019-50339-1 |
| [91] |
Jerde CL, Mahon AR, Chadderton WL et al. "Sight-unseen" detection of rare aquatic species using environmental DNA. Conservation Letters, 2011, 4(2): 150-157. DOI:10.1111/j.1755-263X.2010.00158.x |
| [92] |
Mahon AR, Jerde CL, Galaska M et al. Validation of eDNA surveillance sensitivity for detection of Asian carps in controlled and field experiments. PLoS One, 2013, 8(3): e58316. DOI:10.1371/journal.pone.0058316 |
| [93] |
Kara EL, Hanson PC, Hu YH et al. A decade of seasonal dynamics and co-occurrences within freshwater bacterioplankton communities from eutrophic Lake Mendota, WI, USA. The ISME Journal, 2013, 7(3): 680-684. DOI:10.1038/ismej.2012.118 |
| [94] |
Boere AC, Rijpstra WIC, DE Lange GJ et al. Preservation potential of ancient plankton DNA in Pleistocene marine sediments. Geobiology, 2011, 9(5): 377-393. DOI:10.1111/j.1472-4669.2011.00290.x |
| [95] |
Capo E, Debroas D, Arnaud F et al. Long-term dynamics in microbial eukaryotes communities: A palaeolimnological view based on sedimentary DNA. Molecular Ecology, 2016, 25(23): 5925-5943. DOI:10.1111/mec.13893 |
| [96] |
Foissner W, Berger H. A user-friendly guide to the ciliates (Protozoa, Ciliophora) commonly used by hydrobiologists as bioindicators in rivers, lakes, and waste waters, with notes on their ecology. Freshwater Biology, 1996, 35(2): 375-482. DOI:10.1111/j.1365-2427.1996.tb01775.x |
| [97] |
Ge H, Walhout AJM, Vidal M. Integrating 'omic' information: A bridge between genomics and systems biology. Trends in Genetics, 2003, 19(10): 551-560. DOI:10.1016/j.tig.2003.08.009 |
 2021, Vol. 33
2021, Vol. 33 

