(2: 中国科学院南京地理与湖泊研究所湖泊与环境国家重点实验室, 南京 210008)
(2: State Key Laboratory of Lake Science and Environment, Nanjing Institute of Geography and Limnology, Chinese Academy of Sciences, Nanjing 210008, P. R. China)
蓝藻水华在富营养化湖泊中广泛存在,微囊藻(Microcystis)是形成水华的优势类群,从而受到高度重视,是水华藻类中研究最多的一种[1].它们繁殖快,产生毒素,给生态环境带来了很大的危害[2].微囊藻具有与革兰氏阴性细菌相似的细胞壁[3],具有较强的暗适应和低温适应等生理特点[4],并具有不同的形态种,存在群落结构的演替[4],这些都引起了科学家们越来越多的关注,但是目前其形成水华的机理仍在进一步探索中[5].而从RNA分子水平上研究微囊藻应对环境的响应机制,有助于从微囊藻自身生理特性以及从转录水平上研究揭示水华发生机理[6].
微囊藻大多以群体形式存在于自然水体中,从微囊藻群体中提取高质量的RNA是微囊藻分子生物学研究的重要前提.任何RNA基础分析的技术,如Northern杂交、mRNA纯化、PCR扩增、cDNA合成和文库构建等研究都需要纯度高和完整性好的RNA[7-9].然而,不同的RNA提取方法能够产生不同质量的RNA[10-11].微囊藻群体细胞壁外层的胶鞘较厚,影响RNA提取的物质如蛋白质、酚类等的含量较高,RNase的活性也比较高,导致微囊藻群体RNA的提取困难.同时,微囊藻群体中含有丰富的胞外多糖,使RNA的提取更加困难[12-14],因此是否能够去除多糖等杂质是影响RNA提取质量的关键因素.目前,有许多种方法能够从多糖丰富的样品中提取RNA[13, 15-16].为了选择一个最适合囊藻群体RNA提取的方法,本研究以室内培养的微囊藻群体为材料,对比分析4种针对多糖含量较高的RNA提取方法对微囊藻群体总RNA提取的效果,以期筛选出最适合微囊藻群体RNA的提取方法.
1 材料与方法 1.1 实验材料 1.1.1 藻种及培养采用本实验室自野外分离纯化的铜绿微囊藻(Microcystis aeruginosa)群体D2(由蔡元锋博士在2011年从太湖分离得到),作为RNA提取的样本.采用BG-11培养基[17]在光照培养箱中进行微囊藻群体的培养.光照培养箱温度设为25℃,光暗比为12 h:12 h,光照强度为27 μmol/(m2·s).待藻群体生长到对数生长期,叶绿素a浓度为28.47±2.12 μg/L时,取相同体积(40 ml)藻液若干份,用孔径为3 μm的聚碳酸酯膜(millipore)收集藻群体,-80℃保存,用于RNA提取.
1.1.2 主要仪器与试剂光照培养箱(GZL-P380B,合肥华德利科学器材有限公司),超净工作台(SW-CJ-1FD,苏州安泰),小型高速冷冻离心机(Centrifuge 5415R,Eppendorf公司),电泳仪(DYCP-32B,北京六一仪器厂),实时荧光定量PCR仪(Mastercycler ep Realplex, Eppendorf, Germany),凝胶成像仪(ChemiDoc MP, BIO-RAD),快速核酸提取仪(美国MP FastPrep-24).
FastRNA® Pro Blue Kit试剂盒(美国Mpbio公司)、Qiagen RNeasy Mini Kit试剂盒、溴化乙锭(EB)、琼脂糖、无水乙醇、75 %乙醇、DEPC-treated ddH2O、氯仿、西曲溴铵(CTAB)、β-巯基乙醇(β-ME)和琼脂粉,其它均为分析纯试剂.
1.2 实验方法选取4种针对多糖含量丰富的样品中提取总RNA的方法,分别从含有微囊藻群体的3 μm的聚碳酸酯膜中提取RNA,FastRNA® Pro Blue Kit和Qiagen RNeasy Mini Kit 2种试剂盒法本身包含机械破碎过程,PGTX和CTAB法增加机械破碎过程如下:将样品转移到2 ml裂解管E(Lysing Matrix E, MP Biomedicals,美国)中,反复颠倒混匀,将裂解管E放置于FastPrep-24样品处理仪器中,6 m/s条件下破碎30 s.
1.2.1 方法1PGTX-bead参考Pinto等[13]方法提取RNA,并作出适当改进.具体过程如下:①向样品中加入PGTX(苯酚19.8 g,甘油3.45 ml,8-羟基喹啉0.05 g,乙二胺四乙酸(EDTA)0.29 g,乙酸钠0.4 g,异硫氰酸胍4.75 g,盐酸胍2.3 g,1 ml Triton-X 100,用灭菌的DEPC水定容到50 ml)缓冲液后,使用机械破碎过程和95℃水浴5 min相结合的方法裂解细胞,然后立即冰浴5 min,加入1-溴-3氯丙烷,剧烈涡旋混匀,室温下放置10 min;②离心后转移上清液到新的离心管中,加入等体积的异丙醇沉淀,室温放置10 min后弃上清液;③加入无菌DEPC水配置的75 %的乙醇洗涤,离心后倒掉液体,无菌操作台干燥;④用无菌DEPC水溶解沉淀.
1.2.2 方法2CTAB-bead参考Wang等[15]的方法,并作出适当改进.具体过程如下:①用预热的CTAB(2 % CTAB,2 % PVP,1.4 mol/L NaCl,100 mmol/L Tris-HCl(pH=8.0),20 mmol/L EDTA,1 % β-巯基乙醇(β-ME))缓冲液悬浮样品,涡旋;②进行机械破碎;③裂解液用氯仿/异戊醇(24:1)抽提2次;④吸取上层水相,加入等体积的异丙醇沉淀RNA;⑤用75 %的冷乙醇洗涤沉淀;⑥沉淀在无菌操作台干燥,最后用无菌DEPC水溶解沉淀.
1.2.3 方法3FastRNA® Pro Blue kit主要按照说明书的步骤进行,并做如下改进,具体过程:①用RNApro溶液悬浮收集的藻体,并进行涡旋;②将涡旋后的样品进行机械破碎;③在4℃条件下进行全速离心,转移上清液到新的离心管中;④加入氯仿涡旋10 s,室温静置5 min,再离心,转移上清液到新的离心管中;⑤用氯仿/异戊醇(24:1)进行抽提,转移上清到新的离心管中;⑥加入冷的无水乙醇沉淀RNA,上下颠倒混匀,离心后弃上清;之后用75 %的乙醇洗涤沉淀,最后用无酶无菌水(RNase-free water)溶解沉淀,-80℃条件下保存.
1.2.4 方法4Qiagen RNeasy mini kit主要按照说明书的步骤进行,并做如下改进:①用缓冲液RLT悬浮收集到的藻细胞,涡旋;②进行机械破碎;③在13000转/min、4℃条件下离心15 s,上清液用等体积的70 %乙醇混合,然后将混合物转移到柱子上;④用RW1和RPE 2种缓冲液各冲洗2次;⑤用50 μl无酶无菌水(RNase-free water)溶解沉淀,-80℃条件下保存.
1.2.5 RNA完整性及质量检测取2 μl RNA样品,用1 %的琼脂糖凝胶电泳检测完整性. Nanodrop ND1000分光光度计测定RNA浓度,并测定D260/D280和D260/D230的比值以确定RNA纯度.
1.2.6 荧光定量PCR为验证4种方法提取的RNA样品中DNA污染情况,扩增光合作用相关基因:核酮糖1,5-二磷酸羧化酶/加氧酶(Rubisco,Rbcl),引物序列:上游为5′-CGTTTCCCCGTCGCTTT-3′,下游为5′-CCGAGTTTGGGTTTGATGGT-3′,扩增片段长度128 bp[13, 18],分别对上述4种方法提取的RNA和用DNase处理后的RNA用Mastercycler ep Realplex(Eppendorf, Germany)荧光定量PCR仪和SYBR Premix Ex Taq Ⅱ kit(TaKaRa)进行荧光定量PCR,反应总体积为25 μl,包含1×SYBR Premix Ex TaqTM,0.4 μmol/L的上下游引物和模板.扩增程序:95℃预变性1 min;95℃变性15 s,60℃复性1 min,72℃延伸30 s,共40个循环.
1.2.7 RNA中的污染DNA去除用RNase-free DNaseⅠ(TaKaRa)去除DNA,操作按照说明书的步骤进行,具体过程:①反应体系:RNA 20~50 μg,5 μl 10×DNaseⅠ缓冲液,2 μl DNaseⅠ(RNase-free,5 U/μl),0.5 μl RNase抑制剂(40 U/μl),补水至总体积50 μl;②37℃反应20~30 min;③加入50 μl灭菌的DEPC水,再加入100 μl的苯酚/氯仿/异戊醇(25:24:1),充分混匀;④离心,将上层(水层)移至另一微量离心管,加入100 μl的氯仿/异戊醇(24:1),充分混匀;⑤离心,取上层(水层)移至另一微量离心管,加入10 μl的3 mol/L的乙酸钠(pH 5.2)和250 μl的冷无水乙醇,-20℃条件下放置30~60 min;⑥离心回收沉淀,用70 %的冷乙醇清洗沉淀,真空干燥,用适量无菌DEPC水溶解.
1.2.8 反转录荧光定量PCR去除DNA污染后,根据不同方法提取的RNA浓度不同,计算400 ng所需体积进行反转录.按照Transciptor First Strand cDNA Synthesis Kit说明书的要求进行反转录:加入1 μl Anchored-oligo(dT)和2 μl随机六聚物引物,400 ng RNA和适量PCR级别的水,使总体积为13 μl,混合之后在PCR仪中65℃温浴10 min.温浴之后再依次加入4 μl transcriptor reverse transcriptase reaction缓冲液、0.5 μl RNA抑制剂、2 μl脱氧核苷酸混合剂和0.5 μl transcriptor reverse transcriptase,混匀之后放在PCR仪上反应,设置PCR仪的程序为25℃ 10 min,55℃ 30 min以及85℃ 5 min.将反应完之后的PCR管立即放置冰上,将得到的cDNA保存在-20℃中备用.
PCR过程中以微囊藻特异16S rRNA为内参基因,引物序列为:上游为5′-GCCGCRAGGTGAAAMCTAA-3′,下游为5′-AATCCAAARACCTTCCTCCC-3′,PCR反应条件:95℃预变性10 min; 95℃变性10 s,55℃复性10 s,72℃延伸30 s,共45个循环[19].目的基因为Rbcl,以cDNA为模板,进行PCR扩增,反应体系和扩增程序如上所述(1.2.6).每个样品的内参基因Ct值与目的基因Ct值之差即为ΔCt,用2ΔCt法计算出各个样品相对于内参基因的目的基因的相对表达量[16],然后再进行样品间相对表达量的比较.
2 结果与分析 2.1 PGTX和CTAB法增加机械破碎前、后的RNA提取效果比较为了阐明加入机械破碎对提取微囊藻群体总RNA效果的影响,本文采用FastPrep-24快速核酸提取仪对PGTX和CTAB 2种方法进行机械破碎前后的RNA提取效果比较. PGTX和CTAB 2种方法提取RNA的D260/D280和D260/D230的比值变化不大,表明机械破碎前、后对RNA的纯度影响不大;但PGTX法增加机械破碎后产量大约是破碎前的3倍,CTAB法增加机械破碎后产量大约是破碎前的2倍,表明机械破碎能够显著增加微囊藻群体总RNA的提取产量(图 1).
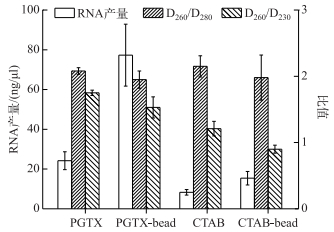
|
图 1 PGTX和CTAB法增加机械破碎前后提取的RNA产量和纯度 Fig.1 Yield and purity of RNA extracted before and after the addition of mechanical disruption of the PGTX and CTAB methods |
采用琼脂糖凝胶电泳检测RNA完整性,结果表明4种方法都能够从微囊藻群体中提取到总RNA(图 2),方法1和方法2电泳条带(23S/16S)清晰明亮,RNA完整性好,但方法2能观察到清晰的DNA条带,DNA污染严重;方法3和方法4电泳条带微弱,结合表 1可知,这2种方法提取的RNA完整性较好,但产量低.
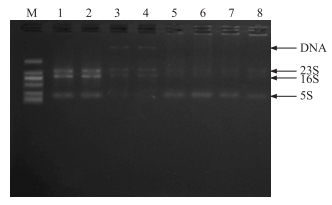
|
图 2 4种方法提取的微囊藻群体RNA琼脂糖凝胶电泳图谱 (1~2:方法1提取的总RNA;3~4:方法2提取的总RNA;5~6:方法3提取的总RNA;7~8:方法4提取的总RNA;M:DL2000 Marker) Fig.2 Agarose gel electrophoresis for RNA extracted from Microcystis colonies by the four methods (1~2:RNA extracted from Method 1;3~4:RNA extracted from Method 2; 5~6:RNA extracted from Method 3;7~8:RNA extracted from Method 4;M:DL2000 Marker) |
| 表 1 4种方法提取的RNA的产量和纯度比较 Tab.1 Comparison of RNA yield and purity obtained by the four methods |
方法1和方法2从微囊藻群体中提取的RNA浓度分别为77.3和15.4 ng/μl,而方法3和方法4提取的RNA浓度仅分别为8.9和7.4 ng/μl(表 1).方法1提取的RNA浓度明显高于其他3种方法(P < 0.05),大约是方法4的10倍. Nanodrop ND1000分光光度计检测结果表明,4种方法获得的RNA样品D260/D280的比值都接近2.0,表明RNA样品中没有蛋白质等杂质的污染;而D260/D230的比值都小于2.2,说明存在酚类和糖类等杂质污染,方法1提取的RNA酚类和糖类等污染较小,方法3污染最严重.
2.4 qPCR检测基因组DNA污染qPCR结果(图 3)可知,方法2提取的RNA样品直接进行荧光定量PCR,Ct值仅为14.34±0.05,DNA污染最严重;方法1和方法4提取的RNA样品直接进行荧光定量PCR,Ct值分别为20.9±0.06和22.92±0.02,DNA污染较小;用DNase处理后,4种方法提取的RNA样品进行荧光定量PCR,Ct值都在30.0左右,与未加模板的阴性对照的荧光定量PCR Ct值接近(30.52±0.24),表明RNA样品中的基因组DNA基本被去除.
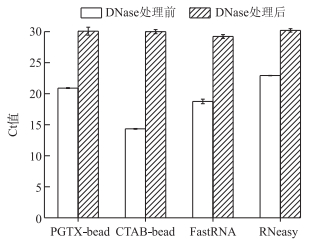
|
图 3 4种方法提取的微囊藻群体总RNA用DNase处理前、后qPCR分析 Fig.3 qPCR analysis of total RNA extracted from Microcystis colonies using the four methods before and after DNase treatment |
qPCR结果表明不同RNA提取方法之间存在差异(表 2).因为16S rRNA更加丰富和稳定,所以qPCR结果受到特定信使RNA检测的高度影响.方法4提取的RNA的Rbcl基因相对表达水平最高,为7.88×10-4,方法1次之,为6.18×10-4,而方法3和方法2提取的RNA的Rbcl基因表达水平都较低(表 2).
| 表 2 4种方法提取RNA的Rbcl基因相对表达量 Tab.2 Relative expression of Rbcl gene using RNA extracted from the four methods |
铜绿微囊藻群体中含有丰富的胞外多糖,通常情况下,胞外多糖以粘液层或胶鞘的形式围绕在群体周围,其粘度值相当于甚至高于黄原胶[20],不仅难以实现完全的细胞溶解,还干扰大多数核酸净化过程[21-22];同时,铜绿微囊藻细胞也含有丰富的核酸内切酶、核酸外切酶和光合色素,它们可以抑制酶促反应,尤其是逆转录[23-24]和PCR[25-26].这些因素的存在使RNA的提取更加困难,严重地降低了RNA的提取产量和纯度. RNA的提取方法有很多,提取过程大致可分为3个部分:细胞裂解、从周围基质中提取RNA和纯化,细胞裂解在提取过程中最为重要[27].提取RNA的基本原理都是将细胞裂解后,把RNA与蛋白质、糖类和DNA等杂质分离开. RNA的纯度和提取量是评价核酸提取方法的一个重要指标,因为提取产量高、纯度好的RNA是进行各种后续研究的前提.因此,根据所用微囊藻群体自身理化性质的不同,选择最适合从微囊藻群体中提取总RNA的方法至关重要.
方法1 PGTX-bead法是一种强变性剂法,PGTX缓冲液能够迅速抑制RNase的活性.苯酚和胍盐是非常有效的蛋白质变性剂,他们的结合使核糖核酸酶快速变性[28-29].同时加入8-羟基喹啉,它既是苯酚稳定剂又是核糖核酸酶抑制剂[30].该方法的优点是蛋白变性剂的存在,能够强烈抑制RNase的活性,可用于大量细菌总RNA的提取.与改进前[13]相比,使用机械破碎和95℃水浴5 min相结合的方法裂解细胞,总RNA提取量明显增加.在4种方法中,方法1提取的RNA质量和产量最高,且成本较低,目的基因表达水平较高,最适合从微囊藻群体中提取总RNA.
方法2 CTAB-bead法,浓度较高的CTAB可沉淀核酸和酸性多聚糖,有效地裂解细菌细胞壁,与β-巯基乙醇共同作用使蛋白变性[31].因此,在含有大量多聚糖的有机体中提取核酸时,可以利用CTAB作为裂解液[32].再与机械破碎法相结合,在FastPrep均质破碎仪中激烈振荡使微囊藻细胞壁及其外层的胶鞘充分裂解,从而有效地提取总RNA.方法2提取的RNA样品凝胶电泳图像(图 1)和qPCR(图 2)结果可知,此方法提取的微囊藻群体总RNA中DNA污染严重,适合需要同时提取DNA和RNA的样本.
方法3 FastRNA® Pro Blue Kit法,是一种能够从革兰氏阴性细菌和革兰氏阳性细菌中快速有效的提取总RNA的试剂盒法. RNApro溶液在细胞裂解过程中使RNase失活,防止RNA降解.从表 1和图 2结果可知,方法3 FastRNA® Pro Blue Kit法提取的微囊藻群体总RNA浓度低,DNA污染较严重,目的基因表达水平很低,而且成本较高,不适合从微囊藻群体中提取RNA.
方法4 Qiagen RNeasy Mini Kit是一种应用比较广泛的RNA提取试剂盒,使用硫氰酸胍盐缓冲液溶解样品,用硅胶膜核酸纯化柱净化RNA.此种试剂盒法提取的微囊藻群体总RNA浓度较低,但DNA污染小,RNA完整性好,目的基因表达水平最高,且操作简单,可以用于从少量微囊藻群体中提取总RNA.
此外,在实验初期,我们也使用了trizol法进行微囊藻群体总RNA提取,但实验结果并不理想.本研究所采用的4种方法中,对PGTX和CTAB 2种方法机械破碎增加前后RNA产量及纯度进行了对比,结果表明,机械破碎能够显著增加RNA产量,同时,另外2种试剂盒在提取步骤中也自带对样品进行机械破碎这一步,说明机械破碎能够使细胞充分裂解,降低微囊藻群体RNA提取难度,增加提取产量,对提取RNA是非常必要的步骤. Nanodrop ND1000分光光度计测定总RNA的D260/D280和D260/D230比值,来衡量RNA纯度.一般D260/D280的比值大约为2.0,表明RNA样品中不存在蛋白质等污染;D260/D230的比值在2.2左右,表明RNA样品中不存在酚类和糖类等污染[15].从实验结果可以看出,4种方法提取的微囊藻群体总RNA都不存在蛋白质等污染,但D260/D230比值都小于2.2,说明存在酚类和糖类等污染.结合微囊藻群体的特点,我们推测,之所以所有方法的D260/D230比值都比较小,可能是因为微囊藻群体中大量胞外多糖的存在,它不仅影响细胞裂解过程,也有极少部分随RNA一起被提取出来.
此外,在凝胶电泳图谱中,CTAB-bead法有明显的DNA条带,说明存在DNA污染,其他3种方法电泳图谱检测不到DNA条带,因此本研究采用了更加灵敏的qPCR方法检测4种方法提取的RNA样品中基因组DNA污染情况及基因组DNA去除情况[11].同时,对去除DNA后的样品进行反转录荧光定量PCR,检测提取的RNA中Rbcl基因的表达水平,结果表明方法1 PGTX-bead法提取的RNA不仅产量高,目的基因表达水平也高;方法4 Qiagen RNeasy Mini Kit法虽然产量低,但目的基因表达水平最高;而方法3 FastRNA® Pro Blue Kit法目的基因表达水平较低.类似不同的RNA提取方法导致目的基因表达水平不同的结果在其他微生物RNA提取过程中也有相关报道[16, 33-34].由此可见,选择合适、统一的RNA提取方法对系统研究微生物的功能的重要性.
4 结论本研究的4种方法,都能够从微囊藻群体中提取到RNA.方法1提取量高、纯度好、成本低,且适合大量提取,是最适合从微囊藻群体中提取RNA的方法.方法2适合于需要同时提取DNA和RNA的样本.方法4是试剂盒提取法,成本高、产量低,但RNA质量较好,且操作简单、耗时短,所检测目的基因的相对表达量高,可用于从少量的微囊藻群体中提取总RNA.由于不同提取方法可能导致目的基因相对表达水平的不同,因此,在同批生物样本的研究中,建议选择统一的RNA提取方法,以减少方法的偏好性带来的误差.
| [1] |
Kong FX, Gao G. Hypothesis on cyanobacteria bloom-forming mechanism in large shallow eutrophic lakes. Acta Ecologica Sinica, 2005, 25(3): 589-595. [孔繁翔, 高光. 大型浅水富营养化湖泊中蓝藻水华形成机理的思考. 生态学报, 2005, 25(3): 589-595.] |
| [2] |
Jiang JL, Song R, Ren JH et al. Advances in pollution of cyanobacterial blooms-producing microcystins and their ecotoxicological effects on aquatic organisms. Progress in Chemistry, 2011, 23(1): 246-253. [姜锦林, 宋睿, 任静华等. 蓝藻水华衍生的微囊藻毒素污染及其对水生生物的生态毒理学研究. 化学进展, 2011, 23(1): 246-253.] |
| [3] |
Gan N, Xiao L, Zhu L et al. The role of microcystins in maintaining colonies of bloom-forming Microcystis spp.. Environmental Microbiology, 2012, 14(3): 730-742. DOI:10.1111/emi.2012.14.issue-3 |
| [4] |
Zhao XX. Thephytoplankton community dynamics and driving factor during bloom period in Taihu Lake[Dissertation]. Hefei: Anhui University, 2013. [赵秀侠. 太湖蓝藻水华形成过程中的浮游植物群落动态及其驱动因素研究[学位论文]. 合肥: 安徽大学, 2013. ]
|
| [5] |
Ma JR, Deng JM, Qin BQ et al. Progress and prospects on cyanobacteria bloom-forming mechanism in lakes. Acta Ecologica Sinica, 2013, 33(10): 3020-3030. [马健荣, 邓建明, 秦伯强等. 湖泊蓝藻水华发生机理研究进展. 生态学报, 2013, 33(10): 3020-3030.] |
| [6] |
Harke MJ, Gobler CJ. Global transcriptional responses of the toxic cyanobacterium, Microcystis aeruginosa, to nitrogen stress, phosphorus stress, and growth on organic matter. PLos ONE, 2013, 8(7): e69834. DOI:10.1371/journal.pone.0069834 |
| [7] |
Murillo I, Raventos D, Jaeck E et al. Isolation of total RNA and mRNA from plant tissues. Promega Notes Mag, 1995, 54: 2-7. |
| [8] |
Hu GC, Honda CH, Kita MY et al. A simple protocol for RNA isolation from fruit trees containing high levels of polysaccharides and polyphenol compounds. Plant Molecular Biology Reporter, 2002, 20(1): 69a-69g. DOI:10.1007/BF02801935 |
| [9] |
Wang D, Douglas D, Kreader C et al. Anintegrated high-throughput system for mRNA purification and quantitation for use in identifying gene knockdown by RNA interference. Journal of the Association for Laboratory Automation, 2006, 11(5): 314-318. DOI:10.1016/j.jala.2006.07.003 |
| [10] |
Nour AM, Barbour EK, Depint F et al. Comparison of five RNA extraction methods from rabbit's blood. Agriculture and Biology Journal of North America, 2010, 1(4): 448-450. DOI:10.5251/abjna.2010.1.4.448.450 |
| [11] |
Rump LV, Asamoah B, Gonzalez-Escalona N. Comparison of commercial RNA extraction kits for preparation of DNA-free total RNA from Salmonella cells. BMC Research Notes, 2010, 3(1): 211. DOI:10.1186/1756-0500-3-211 |
| [12] |
Reynolds C, Jaworski G, Cmiech H et al. On the annual cycle of the blue-green alga Microcystis aeruginosa Kutz. emend. Elenkin. Philosophical Transactions of the Royal Society of London, 1981, 293(1068): 419-477. DOI:10.1098/rstb.1981.0081 |
| [13] |
Pinto F, Thapper A, Sontheim W et al. Analysis of current and alternative phenol based RNA extraction methodologies for cyanobacteria. BMC Molecular Biology, 2009, 10(1): 1-8. DOI:10.1186/1471-2199-10-1 |
| [14] |
Singh S, Sinha R, Daiker V et al. Quantitative and qualitative extraction of RNA from a filamentous cyanobacterium anabaena variabilis PCC 7937. Journal of Applied Phycology, 2010, 22(1): 113-116. DOI:10.1007/s10811-009-9428-7 |
| [15] |
Wang L, Stegemann J. Extraction of high quality RNA from polysaccharide matrices using cetyltrimethylammonium bromide. Biomaterials, 2010, 31(7): 1612-1618. DOI:10.1016/j.biomaterials.2009.11.024 |
| [16] |
Franca A, Melo L, Cerca N. Comparison of RNA extraction methods from biofilm samples of Staphylococcus epidermidis. BMC Research Notes, 2011, 4(1): 572. DOI:10.1186/1756-0500-4-572 |
| [17] |
Allen M, Stanier R. Selective isolation of blue-green algae from water and soil. Journal of General Microbiology, 1968, 51(2): 203-209. DOI:10.1099/00221287-51-2-203 |
| [18] |
Qian H, Yu S, Sun Z et al. Effects of copper sulfate, hydrogen peroxide and N-phenyl-2-naphthylamine on oxidative stress and the expression of genes involved photosynthesis and microcystin disposition in Microcystis aeruginosa. Aquatic Toxicology, 2010, 99(3): 405-412. DOI:10.1016/j.aquatox.2010.05.018 |
| [19] |
Rinta-Kanto J, Ouellette A, Boyer G et al. Quantification of toxic Microcystis spp. during the 2003 and 2004 blooms in western Lake Erie using quantitative real-time PCR. Environmental Science & Technology, 2005, 39(11): 4198-4205. |
| [20] |
Ren XX, Jiang H, Leng X et al. Ecological significance and industrial application of extracellular polysaccharides from cyanobacteria. Chinese Journal of Ecology, 2013, 32(3): 762-771. [任欣欣, 姜昊, 冷欣等. 蓝藻胞外多糖的生态学意义及其工业应用. 生态学杂志, 2013, 32(3): 762-771.] |
| [21] |
Porter RD. DNA transformation. Methods in Enzymology, 1988, 167: 703-712. DOI:10.1016/0076-6879(88)67081-9 |
| [22] |
Wilkins T, Smart L eds. Isolation of RNA from plant tissue. In: Krieg PA ed. A laboratory guide to RNA: Isolation, analysis, and synthesis. New York: Wiley-Liss, 1996: 21-42.
|
| [23] |
Cardellina J, Munro M, Fuller R et al. A chemical screening strategy for the dereplication and prioritization of HIV-inhibitory aqueous natural products extracts. Journal of Natural Products, 1993, 56: 1123-1129. DOI:10.1021/np50097a016 |
| [24] |
Lau A, Siedlecki J, Anleitner J et al. Inhibition of reverse transcription activity by extracts of cultured blue-green algae. Planta Medica, 1993, 59: 148-151. DOI:10.1055/s-2006-959631 |
| [25] |
Cohen M, Wallis J, Campbell E et al. Transposon mutagenesis of Nostoc sp. strain ATCC 29133, a filamentous cyanobacterium with multiple cellular differentiation alternatives. Microbiology, 1994, 140: 3233-3240. DOI:10.1099/13500872-140-12-3233 |
| [26] |
Neilan B. Identification and phylogenetic analysis of toxigenic cyanobacteria using a RAPD PCR. Applied and Environmental Microbiology, 1995, 61: 2286-2291. |
| [27] |
Zhong WH, Wang W, Lin XG et al. Application of nucleic acid-based methods in the study of soil microbial diversity. Acta Pedologica Sinica, 2009, 46(2): 334-341. |
| [28] |
Chomczynski P. A reagent for the single-step simultaneous isolation of RNA, DNA and proteins from cell and tissue samples. Biotechniques, 1993, 15(3): 532-537. |
| [29] |
Chomczynski P, Sacchi N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry, 1987, 162(1): 156-159. |
| [30] |
Emmett M, Petrack B. Rapid isolation of total RNA from mammalian tissues. Analytical Biochemistry, 1988, 174(2): 658-661. DOI:10.1016/0003-2697(88)90069-3 |
| [31] |
Yang ZJ, Gu SQ, Zhang J. Characteristics of several extraction methods on total RNA in plant tissues and strategies to problems. Journal of Anhui Agricultural Sciences, 2009, 37(18): 8341-8342. |
| [32] |
Tan S, Yiap B. DNA, RNA, and protein extraction:The past and the present. Journal of Biomedicine & Biotechnology, 2009, 11: 1-10. |
| [33] |
Franca A, Bento J, Cerca N. Variability of RNA quality extracted from biofilms of foodborne pathogens using different kits impacts mRNA quantification by qPCR. Current Microbiology, 2012, 65(1): 54-59. DOI:10.1007/s00284-012-0124-5 |
| [34] |
Carvalhais V, Delgado-Rastrollo M, Melo L et al. Controlled RNA contamination and degradation and its impact on qPCR gene expression in S. epidermidis biofilms. Journal of Microbiological Methods, 2013, 95(2): 195-200. DOI:10.1016/j.mimet.2013.08.010 |
 2018, Vol. 30
2018, Vol. 30 

