(2: 安徽农业大学生命科学学院, 合肥 230000)
(2: School of Life Science, Anhui Agriculture University, Hefei 230000, P. R. China)
大型溞(Daphnia magna)属于节肢动物门(Arthropoda)、甲壳纲(Crustacea)、鳃足亚纲(Branchiapoda)、枝角目(Cladocera)、溞科(Daphniidae)、溞属(Daphnia), 是微甲壳类浮游动物的重要组成物种[1], 主要生活在淡水中, 具有分布广、生活周期短、繁殖快、易于在实验室培养且对水环境中多种有毒化学物质敏感性强[2]等特点, 因此被广泛应用于水质监测和水生生物毒理研究[3-4], 也是生命科学和环境科学等研究领域较为理想的模式生物[5].目前关于大型溞的研究仍然是毒理研究和水质监测占主导[6-10], 关于溞类的分子进化研究也是一个热点.
动物线粒体基因组由于具有分子量小、含量多、结构简单、进化速率快、母系遗传等特点, 常被用于动物界的分类鉴定、物种多样性和不同等级阶元系统发育的研究[11-14], 它包含丰富的分子标记, 如单个基因序列、基因重排模型和RNA二级结构等, 被广泛应用于种群遗传学、物种鉴定、不同分类阶元的系统发育等研究[15-16].
到目前为止, 溞属浮游动物中只有来自北美的蚤状溞(D. pulex)线粒体DNA全序列已被测定分析并公布[17], 而对另一种溞属浮游动物—大型溞在分子生物学方向的研究尚属少见, 并且溞属动物依据形态学标准进行分类的方法有很多不足, 出现了同名异种或者同种异名现象[18].耿雪侠等[19]基于3种溞属动物线粒体基因组COⅠ基因的测定与分析并构建系统进化树, 结果表明来自中国的隆线溞(D. carinata)和大型溞具有较近的亲缘关系, 而与北美蚤状溞亲缘关系相对较远, 因此需要选择更多的分子标记来进一步验证这个结果并揭示其真正原因.基于此, 本文对大型溞线粒体基因组全序列进行测定和注释, 并对其基因组成和结构特征进行初步分析, 以期为溞科的分类学和系统进化研究提供新的分子生物学数据资料.
1 材料与方法 1.1 材料实验用的大型溞(Daphnia magna)采自淮河.连续培养3代以上的单克隆品系用于基因组DNA的提取.
1.2 基因组DNA的提取将经饥饿处理的单只大型溞, 置于1.5 ml离心管中, 大型溞基因组DNA的提取参照文献[20], 并置于-20℃保存备用.
1.3 PCR扩增及序列测定根据Genbank中已公布的北美蚤状溞(Daphnia pulex)的线粒体基因组全序列(Genbank登录号:AF117817.1)[17]设计出可覆盖大型溞线粒体基因组全序列的引物(表 1).扩增片段间均有部分重叠.
| 表 1 扩增大型溞线粒体基因组全序列的引物 Tab.1 Primers used for amplifying the complete mitochondrial genome sequence of Daphnia magna |
PCR反应总体积为25 μl, 反应体系为:dNTP Mixture (10 mmol/L) 1 μl、10×PCR buffer缓冲液(Mg2+ plus)2.5 μl、DMSO 2 μl、DNA模板1 μl、正反向引物(10 μmol/L)各1 μl、LA-Taq DNA聚合酶(5 U/μl)0.25 μl, 补加16.25 μl ddH2O至25 μl.
PCR反应条件为:94℃预变性5 min; 94℃变性30 s、50℃ 30 s、72℃ 2~8 min, 30个循环; 最后72℃充分延伸10 min, 4℃保存.取5 μl PCR扩增产物进行琼脂糖凝胶电泳检测并拍照. PCR产物经割胶纯化, 产物委托上海美吉生物医药科技有限公司测序.
1.4 序列分析利用DNAStar软件中的SeqMan和测序峰图结果分析软件Chromas 1.62将所得序列进行确认、拼接和组装, 最终获得大型溞线粒体基因组的全序列(Genbank登录号:KP296147).与北美蚤状溞线粒体基因组全序列比对后, 运用在线软件DOGMA(http://dogma.ccbb.utexas.edu/)和MITOS(http://mitos.bioinf.uni-leipzig. de)等对大型溞线粒体基因组进行基因定位, 并预测tRNA基因的二级结构.采用MEGA 6.02[21]软件分析基因组各部分的碱基组成、蛋白质编码基因密码子使用频率、密码子相对使用频率(RSCU)等.
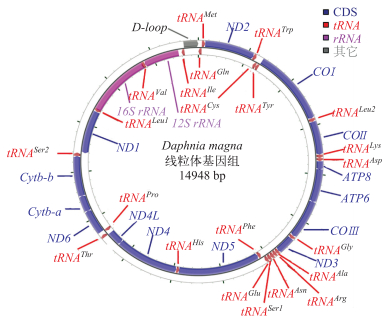
2 结果 2.1 大型溞线粒体基因组组成与基因排列大型溞线粒体基因组全长14948 bp, 包括13个蛋白质编码基因(Protein-coding genes, PCGs)、22个tRNA基因、2个rRNA基因(16S rRNA和12S rRNA)和1个介于tRNAIle基因和tRNAGln基因之间的非编码区(D-loop区)(图 1).其中, 23个基因由J链编码, 另外的15个基因由N链编码.大型溞线粒体基因组的基因结构较为紧凑, 除控制区外, 基因间非编码间隔区仅9处81 bp, 间隔长度为1~62 bp不等, 最大的基因间隔为62 bp, 在12S rRNA和tRNAIle基因之间.同时, 部分相邻基因间存在重叠现象, 共计13处77 bp, 重叠长度在1~27 bp之间, 最长的基因重叠出现在16S rRNA和tRNAVal基因之间, 为27 bp(表 2).
|
图 1 大型溞线粒体基因组组织结构(COⅠ、COⅡ、COⅢ分别表示细胞色素氧化酶亚基Ⅰ~Ⅲ; ATP8和ATP6分别表示ATP合成酶亚基8和6; ND1~ND6和ND4L分别表示NADH脱氢酶亚基1~6和4L; Cytb表示细胞色素b; 12S rRNA和16S rRNA; tRNA基因用氨基酸代码表示) Fig.1 Circular map of the mitochondrial genome of Daphnia magna |
| 表 2 大型溞线粒体基因组组成* Tab.2 Organization of the D. magna mitogenome |
大型溞线粒体基因组中含腺嘌呤(A)4839 bp、鸟嘌呤(G)2329 bp、胸腺嘧啶(T)5192 bp、胞嘧啶(C)2588 bp, 分别占全长的32.37%、15.58%、34.73%、17.31%, A+T占67.11%, G+C占32.89%, A+T含量明显高于G+C含量(表 3), 符合无脊椎动物线粒体DNA序列碱基组成的特点.
| 表 3 大型溞线粒体基因组不同区域的碱基组成* Tab.3 Nucleptide composition in different regions of the D. magna mitochondrial |
大型溞线粒体基因组所包含的13个蛋白质编码基因总长度为10582 bp, 除COⅠ基因以CTG、ATP8基因以GTG、ND3基因以ATC和ND6基因以ATT为起始密码子以外, 其余9个蛋白质编码基因均以ATG作为起始密码子. 9个蛋白质编码基因具有典型的完全终止子TAG或TAA, COⅠ、COⅡ、ND4和ND5基因则以T作为终止信号.在这13个蛋白质编码基因中, ND1、ND4、ND4L和ND5由N链编码, 其余9个基因均由J链编码.蛋白质编码基因没有内含子, 相邻基因之间出现重叠现象, 如ATP6和COⅢ基因间重叠1 bp, ND4和ND4L基因间重叠1 bp(表 2). Cytb基因在10513位置缺失1个碱基T, 在翻译时, 可以通过RNA编辑过程中尿苷酸的添加来形成完整的细胞色素b mRNA序列, 进而翻译成有功能的蛋白质.
大型溞线粒体基因组不同蛋白质编码基因的碱基组成见表 3. 13个蛋白质编码基因的平均A+T含量为66.18%, 其中, 密码子第1、2、3位点的A+T含量基本一致.大型溞线粒体基因组各个不同区域的A+T含量均高于G+C含量, 除tRNA和rRNA基因外, 它们的AT skew均为负值, 呈现出一致的A/T碱基使用偏向性(表 3).
运用MEGA 6.02软件对这13个蛋白质编码基因进行统计分析, 得出总共有3526个密码子(不包含4个不完全的终止密码子), 其中NNU和NNA密码子的RSCU值均大于等于1, 表明其第3位点全部是A或U, 该密码子使用的偏向性与蛋白质编码基因第3位点A+T(66.6%)偏向性有一定的正相关性.
2.3 tRNA基因和rRNA基因利用DOGMA(http://dogma.ccbb.utexas.edu/)和MITOS(http://mitos.bioinf.uni-leipzig.de/index.py)等软件及人工辅助校正得到大型溞线粒体基因组的22个tRNA基因的准确位置和二级结构的预测结果, 基因长度为62~72 bp不等.其中13个由J链编码, 其余的由N链编码.除tRNASer1外, 其余的tRNA均能形成典型的三叶草结构(图 2). tRNASer1缺少二氢尿嘧啶臂(DHU stem), 在其相应位置上只形成1个简单环.除典型的A·U, G·C配对外, 在大型溞的17个tRNA基因中还发现了25对错配, 其中10对在氨基酸臂上, 5对在DHU臂上, 4对在反密码子臂上, 6对在TϕC臂上, 主要是G·U和U·G错配.

|
图 2 大型溞22个tRNA基因二级结构 Fig.2 Predicted clover-leaf secondary structures for 22 tRNA genes of D. magna |
rRNA基因的位置相对固定,在无脊椎动物线粒体中比较保守.大型溞线粒体的16S rRNA基因位于tRNALeu1基因和tRNAVal基因之间, 12S rRNA基因位于tRNAVal基因和tRNAIle基因之间(图 1). 16S rRNA和12S rRNA基因长度分别为1373和752 bp.
2.4 D-loop区大型溞mtDNA的D-loop区位于tRNAIle基因和tRNAGln基因之间(图 1), 全长289 bp, A+T含量为73.70%, 高于线粒体基因组其他区域的A+T含量(表 3), 因此, 也称A+T丰富区.该区不编码蛋白质, 但对基因的转录和复制起着调控作用, 所以又称为控制区.
3 讨论大型溞线粒体基因组全序列长度为14948 bp.通过分析发现, 基因组成与典型节肢动物线粒体基因组一致, 但排列顺序略有差别(图 1).大型溞tRNAIle基因由N链编码, 位置在14596~14659 bp处, 而北美蚤状溞的tRNAIle由J链编码, 在1~64 bp处.其余各基因的位置与北美蚤状溞类似.
在溞类线粒体蛋白质编码基因中, 至少有3种不同的起始密码子(ATG、ATT和GTG), 在其他动物线粒体DNA中也普遍存在[22-23].大型溞COⅠ基因以CTG作为起始密码子, 而北美蚤状溞以ATTA起始.已报道过的COⅠ基因的起始密码子有三联体密码子、四联体密码子和六联体密码子[24]等.目前, 关于COⅠ基因起始密码子的多样性机理尚不明确, 仍需做进一步研究来检测验证.在大型溞13个蛋白质编码基因中, 9个基因以完全终止密码子(TAA和TAG)作为终止信号, 只有COⅠ、COⅠ、ND5和ND4基因使用不完全终止密码子T.而在北美蚤状溞中ND2基因也使用不完全终止密码子T, ATP6和ND3基因则使用另一个不完全终止密码子TA.不完全终止密码子可能通过mRNA加工过程中的多聚腺苷酸化形成完全终止密码子[25-27].
大型溞的tRNASer1缺少二氢尿嘧啶臂(DHU stem), 在欧洲蚤状溞里也发现该现象, 这在后生动物中普遍存在.其他21个tRNA均形成了典型的三叶草结构. 17个tRNA基因共有25对G·U或U·G错配. A+T含量68.79%, 远高于G+C含量.
大型溞16S rRNA和12S rRNA基因长度分别为1373 bp和752 bp, 两者长度与已公布的北美蚤状溞的16S rRNA(1314 bp)和12S rRNA(753 bp)基因长度相似. 16S rRNA和12S rRNA基因的A+T含量分别为71.30%和69.15%, 与北美蚤状溞的16S rRNA(68.19%)和12S rRNA(67.20%)基因的A+T含量基本相似.
大型溞D-loop区长度为289 bp, A+T含量73.70%, 高于线粒体基因组其他区域.北美蚤状溞D-loop区长689 bp, A+T含量67.05%.在脊椎动物和无脊椎动物中, 该区包含复制起始点. D-loop区因不受编码限制, 因此含有丰富的变异位点, 且该区长度的差异是导致整个基因组全序列长度多态性的主要原因.
| [1] |
Forrò L, Korovchinsky NM, Kotov AA et al. Global diversity of cladocerans (Cladocera; Crustacea) in freshwater. Hydrobiologia, 2008, 595: 177-184. DOI:10.1007/s10750-007-9013-5 |
| [2] |
苏乃洲. 氯化铝处理镉污染水体对大型溞(Daphnia magna)毒性效应研究[学位论文]. 济南: 山东师范大学, 2013.
|
| [3] |
Dietrich S, Ploessl F, Bracher F et al. Single and combined toxicity of pharmaceuticals at environmentally relevant concentrations in Daphnia magna-A multigenerational study. Chemosphere, 2010, 79(1): 60-66. DOI:10.1016/j.chemosphere.2009.12.069 |
| [4] |
Fan W, Wu C, Zhao C et al. Application of enriched stable isotope technique to the study of copper bioavailability in Daphnia magna. Journal of Environmental Science, 2011, 23(5): 831-836. DOI:10.1016/S1001-0742(10)60483-4 |
| [5] |
Miner BE, Knapp RA, Colbourne JK et al. Evolutionary history of alpine and subalpine Daphnia in western North America. Freshwater Biology, 2013, 58(7): 1512-1522. DOI:10.1111/fwb.2013.58.issue-7 |
| [6] |
丁婵娟. 铜、铅、锌对大型溞毒性效应研究[学位论文]. 济南: 山东师范大学, 2013.
|
| [7] |
杨晓凡, 陆光华, 刘建超等. 环境相关浓度下的药物对大型蚤的多代慢性毒性. 中国环境科学, 2013, 33(3): 538-545. |
| [8] |
李华, 李秀环, 张大侠等. 不同剂型高效氯氟氰菊酯对大型溞的毒性效应. 环境科学学报, 2014, 34(6): 1615-1620. |
| [9] |
马金奉, 邓春生, 张广恪等. 利用藻-溞系统去除富营养水体中磷的研究. 中国农业科技导报, 2014, 16(1): 148-156. |
| [10] |
王鑫. 不同温度下大型溞(Daphnia magna)抑藻净水效应的研究. 中国农学通报, 2014, 30(2): 105-109. DOI:10.11924/j.issn.1000-6850.2013-2521 |
| [11] |
Cameron SL, Lambkin CL, Barker SC et al. A mitochondrial genome phylogeny of Diptera:whole genome sequence data accurately resolve relationships over broad time scales with high precision. Systematic Entomology, 2007, 32(1): 40-59. DOI:10.1111/j.1365-3113.2006.00355.x |
| [12] |
Ballard JW. Comparative genomics of mitochondrial DNA in members of the Drosophila melanogaster subgroup. Journal of Molecular Evolution, 2000, 51(1): 64-75. DOI:10.1007/s002390010067 |
| [13] |
Buckley TR, Simon C, Chambers GK. Phylogeography of the New Zealand cicada Maoricicada campbelli based on mitochondrial DNA sequences:ancient clades associated with cenozoic environmental change. Evolution, 2001, 55(7): 1395-1407. DOI:10.1111/evo.2001.55.issue-7 |
| [14] |
吴孝兵, 王义权, 周开亚. 扬子鳄的线粒体全基因组与鳄类系统发生. 科学通报, 2003, 48(18): 1954-1958. DOI:10.3321/j.issn:0023-074X.2003.18.011 |
| [15] |
Foottit RG, Maw HEL, Von Dohlen CD et al. Species identification of aphids (Insecta:Hemiptera:Aphididae) through DNA barcodes. Molecular Ecology Resources, 2008, 8: 1189-1201. DOI:10.1111/men.2008.8.issue-6 |
| [16] |
Wei SJ, Min S, Sharkey MJ et al. Coparative mitogenomics of Braconidae (Insecta:Hymenoptera) and the phylogenetic utility of mitochnodrial genomes with special reference to Holometabolous insects. BMC Genomics, 2010, 11: 371. DOI:10.1186/1471-2164-11-371 |
| [17] |
Crease TJ. The complete sequence of the mitochondrial genome of Daphnia pulex (Cladocera:Crustacea). Gene, 1999, 233(1/2): 89-99. |
| [18] |
徐敏. 七种枝角类分子系统分类的初步研究[学位论文]. 淮北: 淮北师范大学, 2014.
|
| [19] |
耿雪侠, 张立, 徐敏等. 3种溞属动物线粒体COⅠ基因及其侧翼序列的PCR扩增与分析. 南京农业大学学报:自然科学版, 2014, 37(3): 44-50. |
| [20] |
彭树英, 邓道贵, 金显文. 单只大型溞DNA的提取及COⅠ基因部分序列的测定分析. 淮北师范大学学报:自然科学版, 2011, 32(3): 40-43. |
| [21] |
Tamura K, Stecher G, Peterson D et al. Mega 6:Molecular evolutionary genetics analysis version 6. 0. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [22] |
杨超, 汪青雄, 黄原等. 棕头鸥线粒体基因组全序列测定与分析. 遗传, 2012, 34(11): 1434-1446. |
| [23] |
夏靖, 胡静, 朱国萍等. 大卫绢蛱蝶线粒体基因组全序列测定和分析. 昆虫学报, 2011, 54(5): 555-565. |
| [24] |
毛增辉, 郝家胜, 朱国萍等. 菜粉蝶线粒体基因组全序列测定和分析. 昆虫学报, 2010, 53(11): 1295-1304. |
| [25] |
Ojala D, Merkel C, Gelfand R et al. The tRNA genes punctuate the reading of genetic information in human mitochondrial DNA. Cell, 1980, 22: 393-403. DOI:10.1016/0092-8674(80)90350-5 |
| [26] |
Moritz C, Dowing TE, Brown WM. Evolution of animal mitochondrial DNA:relevance for population biology and systematic Annual Review of Ecology and Systematics, 1987, 18:269-292.
|
| [27] |
鲁成, 刘运强, 廖顺尧等. 家蚕线粒体基因组全序列测定与分析. 农业生物技术学报, 2002, 10(2): 163-170. |
 2016, Vol. 28
2016, Vol. 28 

